2. Conceptos básicos y herramientas
2.1. Células y microbios: los primeros descubrimientos
Las primeras observaciones
La gran mayoría de las células son microscópicas, es decir, hace falta un microscopio para poder observarlas. Sin embargo, hay otras, como los huevos de aves, que alcanzan varios centímetros de diámetros. El primero en observar células (y en llamarlas así) fue el científico inglés Robert Hooke, quien, en 1665 y usando un microscopio primitivo, describió y dibujó las células muertas de una lámina de corcho.

Pero el primero en observar células vivas fue el holandés Antony van Leeuwenhoek (1632-1723). Para sus observaciones construyó una serie de microscopios rudimentarios pero muy útiles para la época, que le permitieron describir no sólo bacterias, sino también protozoarios de vida libre y parásitos, espermatozoides, células de la sangre y algas.
Su trabajo, aunque meramente descriptivo, descubrió el fabuloso mundo microscópico que hasta ese entonces era totalmente desconocido. Hoy se definen como microorganismos o microbios a aquellos organismos vivos que por su tamaño no pueden verse si no es con la ayuda un microscopio.
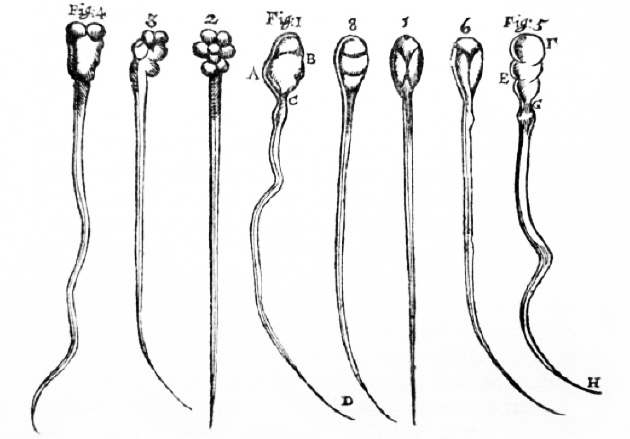
Espermatozoide, tal como los observó Antony van Leeuwenhoek
- Trabajo de Antony van Leeuwenhoek (inglés)
- Cuaderno para docentes Nº 80: cómo se estudian las células
La controversia sobre la generación espontánea
Pero, ¿cuál era el origen de los “animáculos” descritos por Antony van Leeuwenhoek? Luego de sus descubrimientos, la especulación sobre el origen de los microorganismos dividió a los científicos en dos grupos. Por un lado, estaban los que pensaban que los microorganismos provenían de la descomposición de las plantas o animales, es decir, eran el resultado y no la causa de la descomposición. Los que apoyaban esta teoría creían que la vida se generaba a partir de materia no viva, proceso que se denominó abiogénesis y que fue la base del concepto de la generación espontánea. Por otro lado, estaban los que apoyaban la teoría de la biogénesis, que creían que los microorganismos se originaban a partir de otros microorganismos, como ocurre con las plantas y los animales. Aunque hoy resulta obvio que no existe la generación espontánea, llevó más de cien años (¡y muchísimos experimentos!) resolver esta controversia. La idea de la generación espontánea se remonta a los antiguos griegos, quienes creían que los gusanos crecían del lodo. El italiano Francesco Redi demostró en 1668 que los gusanos encontrados en la carne podrida eran las larvas que provenían de los huevos que habían depositado en la carne las moscas y no el producto de la generación espontánea. Pero en 1745 John Needham hirvió trozos de carne para destruir los organismos preexistentes y los colocó en un recipiente abierto. Al cabo de un tiempo observó colonias de microorganismos sobre la superficie y concluyó que se generaban espontáneamente a partir de la carne. En 1769, Lazzaro Spallanzani repitió el experimento, esta vez tapando los recipientes; no aparecieron colonias, lo que contradecía la teoría de la generación espontánea. Pero Needham argumentó que el aire era esencial para la vida y este aire había sido excluido en los experimentos de Spallanzani. Después de muchos experimentos, interpretaciones y discusiones, fue el químico francés Luís Pasteur quien en la década de 1860 terminó con la controversia. A partir de sus propios trabajos sobre la fermentación, a Pasteur le resultaba obvio que las levaduras y otros microorganismos encontrados durante la fermentación y la putrefacción provenían del exterior, del aire. Observando el fenómeno de fabricación del vino, demostró que la fuente de las levaduras era la propia piel de las uvas, que estaba en contacto con el aire. Si extraía estérilmente el jugo de las uvas, este jugo no fermentaba y no se encontraban levaduras. Por otro lado, si cubría con lienzos estériles las uvas desde la cosecha, tampoco crecían levaduras en estas uvas y no se producía el vino. Pero el experimento crucial fue el que realizó en 1864 utilizando frascos (matraces) con un tubo largo y curvado llamados "matraces cuello de cisne". El jugo de uva se colocaba en el frasco y luego de la esterilización el largo cuello del matraz se sellaba en la punta. Si se abría esa punta, el aire pasaba libremente a través del cuello, pero los microorganismos no aparecían en la solución ya que las partículas de polvo y microorganismos sedimentaban en el recodo del cuello. Pero si el jugo de uva tocaba el recodo contaminado, el crecimiento de los microorganismos era inmediato.
La teoría celular
Alrededor de 1833 el botánico escocés Robert Brown descubrió el núcleo celular. Estudiando bajo el microscopio a los tejidos vegetales de diferentes plantas vio que cada célula tenía una zona central más oscura, a la que llamó primero “areola” y luego “núcleo”. Años más tarde, el alemán Matthias Schleiden descubrió que todas las plantas estaban compuestas por células, y, un año después, su compatriota Theodor Schwann arribó a la misma conclusión, pero con los animales. Así, sentaron las bases de lo que luego sería la “Teoría Celular”, que dice que todos los organismos vivos están formados por una o más células. Esta teoría fue luego completada por Rudolph Virchow, quien concluyó en 1855 que toda célula proviene de una existente. Hoy la teoría celular se basa en los siguientes postulados:
- Todos los seres vivos están formados por células
- Todas las células derivan de otra preexistente
- Todas las funciones vitales de los organismos ocurren dentro de las células
- Las células contienen la información necesaria para realizar sus propias funciones y las funciones de las próximas generaciones de células.
2.2. Características de las células - células eucariontes y procariontes
Hay organismos formados por una única célula (unicelulares), como las bacterias, las levaduras y las amebas. Hay otros más complejos, formados por muchas células (pluricelulares), como las plantas y animales, por ejemplo. En estos organismos, las células se ordenan en tejidos, los que su vez forman los órganos. Aunque pueden tener formas, tamaños y funciones diferentes, todas las células comparten características muy importantes:
- Están rodeadas de una membrana celular o plasmática que las separa del exterior, pero a la vez permite el intercambio con el medio externo. Algunas células, como las bacterias y las células de hongos y plantas, presentan una pared celular por fuera de la membrana plasmática.
- La membrana plasmática rodea al citoplasma, una solución acuosa viscosa donde están inmersas las organelas y donde ocurren importantes procesos metabólicos.
- El material genético o hereditario de todas las células es el ADN, o ácido desoxirribonucleico.
- Metabolismo: Las células se alimentan por sí mismas, toman los nutrientes del medio, los transforman en otras moléculas, producen energía y excretan los desechos de estos procesos.
- Reproducción: las células se originan por división de otras células.
- Diferenciación: durante el desarrollo de los organismos pluricelulares muchas células pueden cambiar de forma y función, diferenciándose del resto. La diferenciación celular hace que una célula comience a fabricar algo que antes no fabricaba y esto está asociado a una función particular. Una neurona, por ejemplo, es una célula especializada en la transmisión del impulso nervioso.
- Señalización química. Las células responden a estímulos químicos y físicos y suelen interactuar y comunicarse entre sí, como ocurre en los organismos pluricelulares complejos a través de las hormonas, los neurotransmisores y los factores de crecimiento.
Si bien todas las células comparten las características mencionadas más arriba, presentan una serie de diferencias que permiten agruparlas en dos grandes categorías: procariontes y eucariontes. Las células procariontes no tienen núcleo ni organelas (estructuras celulares rodeadas de membrana, como las mitocondrias, los cloroplastos, los lisosomas, etc.) y su organización interna es simple. Su material genético se encuentra formando un único cromosoma circular. Las células procariontes son pequeñas, de 0,1 a 3 micrones (un micrón es la milésima parte de un milímetro) y forman parte de organismos unicelulares que viven solitarios o en colonias. A estos seres se los llaman organismos procariontes, y son las bacterias y las cianobacterias (algas verdeazules). Se reproducen por un mecanismo simple, conocido como fisión binaria, en el que el material genético se duplica y luego la célula se divide en dos células hijas iguales.
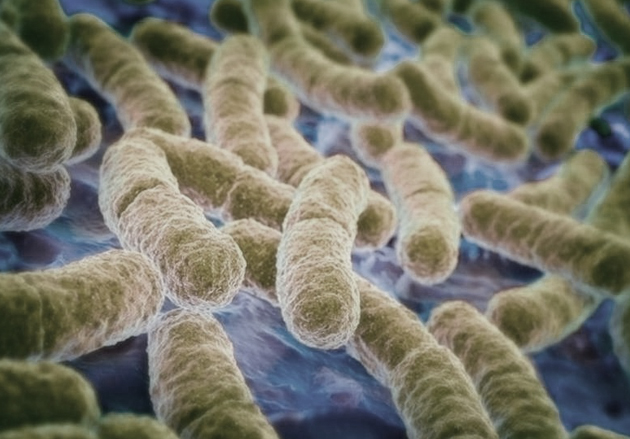
Bacterias Escherichia coli
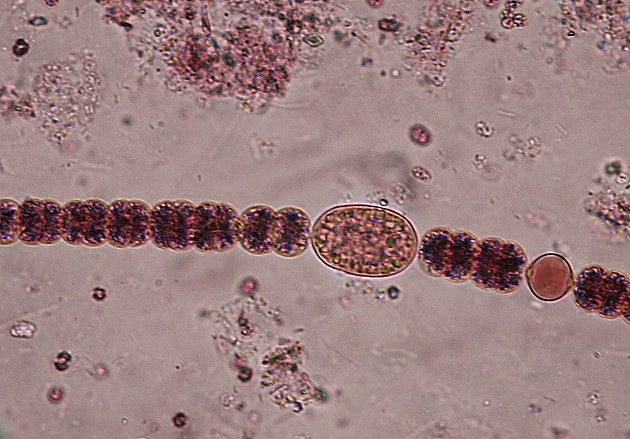
Cianobacteria Anabaena sphaerica
Las células eucariontes, en cambio, tienen núcleo y organelas, y su organización interna es más compleja. Son más grandes que las procariontes, tienen entre 2 y 100 micrones. Su material genético se encuentra distribuido en varios cromosomas lineales. Se dividen por un mecanismo especial y coordinado llamado mitosis, que asegura la correcta distribución del material genético entre las células hijas y el mantenimiento del número de cromosomas de la especie. Las células eucariontes forman parte de organismos unicelulares o pluricelulares. Estos seres son los hongos, protozoarios, plantas y animales.
- Cuaderno para docentes Nº 50: las especies modelos en biotecnología
2.3. Composición de las células
El agua, los iones inorgánicos y las moléculas orgánicas pequeñas constituyen aproximadamente el 75-85% del peso de la materia viva. De todas estas moléculas, el agua es de lejos la más abundante. El resto está compuesto por moléculas más grandes, denominadas “macromoléculas”, que son las proteínas, polisacáridos, lípidos y ácidos nucleicos.
Composición aproximada de los componentes de una célula bacteriana
Componente |
% del peso total de la célula |
|
agua |
70 |
|
iones inorgánicos |
1 |
|
azúcares |
1 |
|
aminoácidos |
0,5 |
|
nucleótidos |
0,5 |
|
ácidos grasos |
1 |
|
macromoléculas (proteínas, ácidos nucleicos, lípidos y polisacáridos) |
26 |
Las células contienen cuatro tipos principales de moléculas orgánicas pequeñas: azúcares, ácidos grasos, nucleótidos y aminoácidos. Se las puede encontrar libres en el citoplasma o dentro de alguna organela, vesícula o membrana, donde participan de diferentes procesos. Algunas pueden ser transformadas en moléculas más pequeñas aún, con el fin de que la célula obtenga la energía necesaria para sus funciones. Además, la mayor parte de estas moléculas pequeñas son usadas como “ladrillos” (monómeros) para construir enormes macromoléculas (polímeros): las proteínas, ácidos nucleicos, lípidos y polisacáridos.
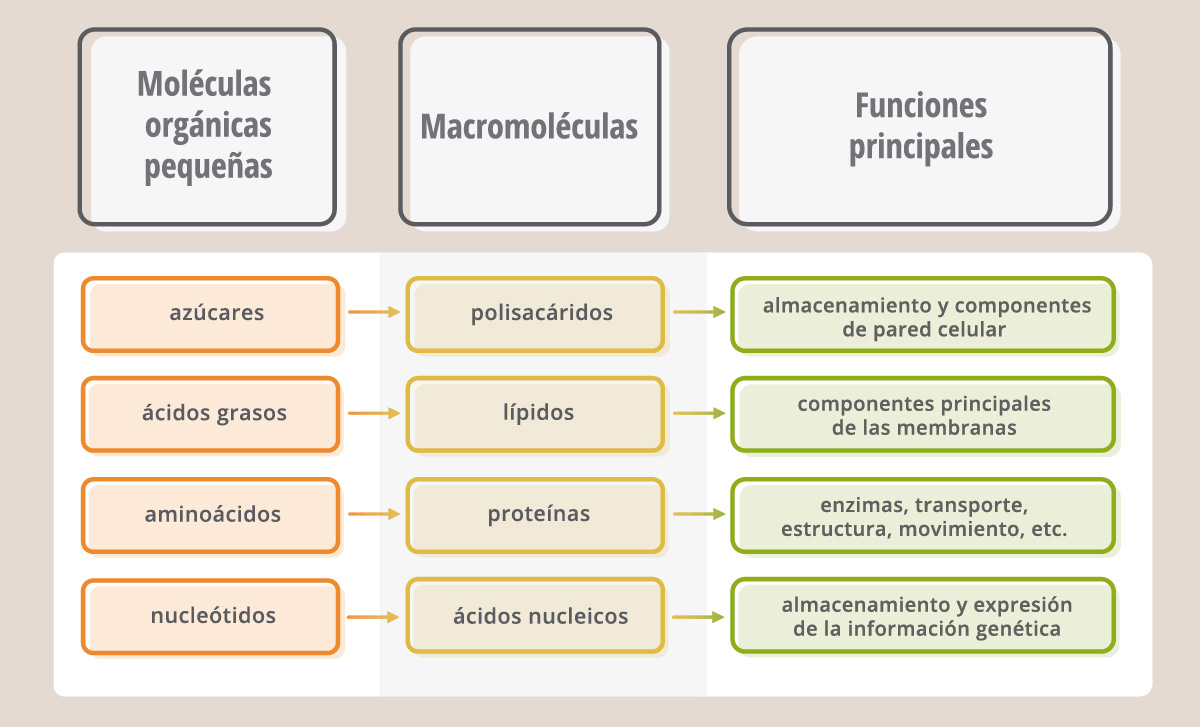
Las macromoléculas, y en particular las proteínas y los ácidos nucleicos, son los componentes más interesantes y característicos de los sistemas vivientes.
Las proteínas
Las proteínas son las verdaderas obreras de la célula, y también las macromoléculas más abundantes y diversas en estructura y función. Un hepatocito (célula del hígado) tiene unas 10.000 proteínas diferentes, ¡y cada una se encuentra repetida aproximadamente un millón de veces! Hay proteínas estructurales, como las que le dan forma a la célula, otras transportan oxígeno, como la hemoglobina, otras participan en la respuesta inmune contra los agentes patógenos, como los anticuerpos. Pero muchas son enzimas, proteínas que tienen la capacidad de acelerar (catalizar) reacciones químicas que no podrían ocurrir espontáneamente en la célula. Sin las enzimas, los procesos celulares como la reproducción, conversión de alimento en energía, construcción de macromoléculas, excreción de desechos celulares, entre otros, no serían posibles.
Las proteínas son polímeros de aminoácidos.
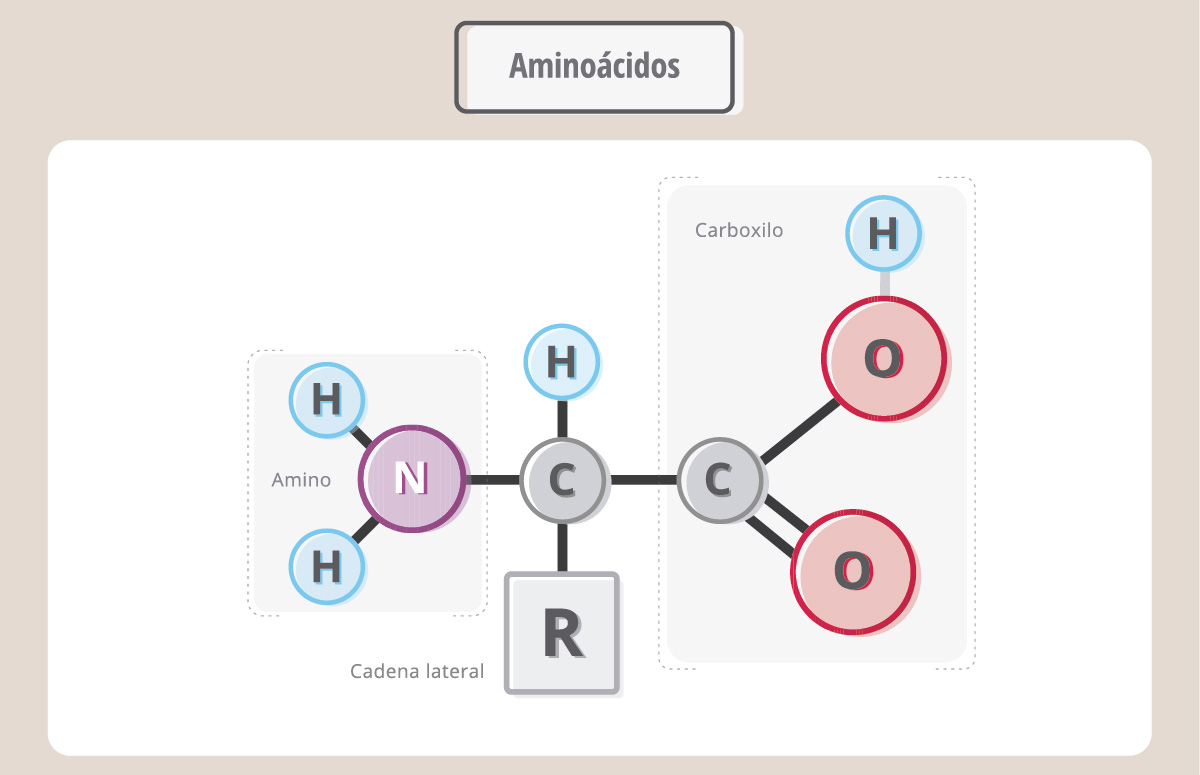
Hay 20 aminoácidos diferentes, pero todos ellos tienen una fórmula básica común, constituida por un carbono central al que se le unen un grupo químico carboxilo, uno amino y otro grupo químico que es particular para cada aminoácido y que se conoce como “cadena lateral o R”. Para formar una proteína, los aminoácidos se unen uno tras otro a través de una unión covalente particular, denominada “unión peptídica”, que involucra al grupo carboxilo de un aminoácido y al amino del siguiente.
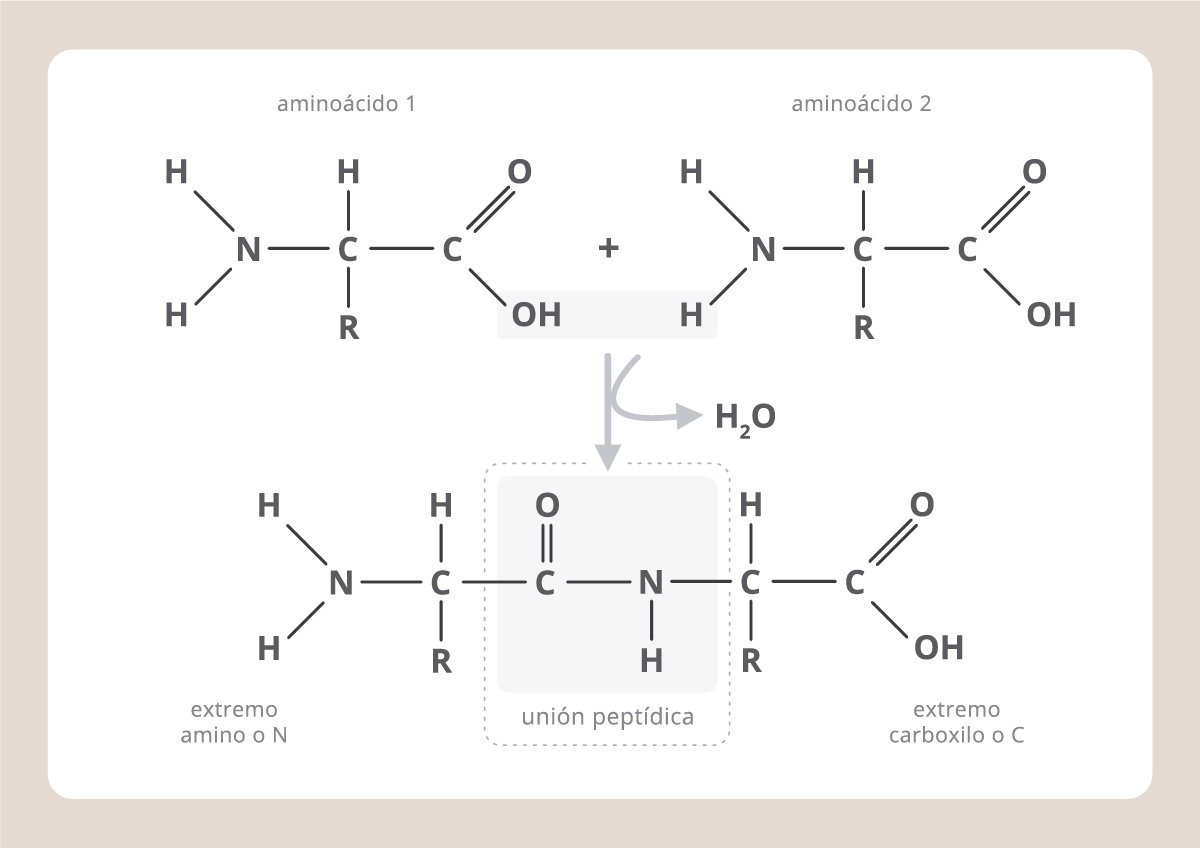
La sucesión particular de aminoácidos en una proteína determina su “estructura primaria”, donde los aminoácidos se encuentran como cuentas en un collar. Pero las características de los grupos laterales de los aminoácidos hacen que éstos, aunque se encuentren alejados en el collar, puedan acercarse en el espacio. Así, la proteína adopta una conformación tridimensional (estructuras secundaria y terciaria) que es propia de cada proteína, ya que este plegamiento depende de la secuencia de aminoácidos, y cada proteína tiene una secuencia particular. Finalmente, varias cadenas proteicas plegadas pueden unirse entre sí por uniones no covalentes, constituyendo la estructura cuaternaria, como en el caso de la hemoglobina, que está formada por cuatro subunidades iguales.
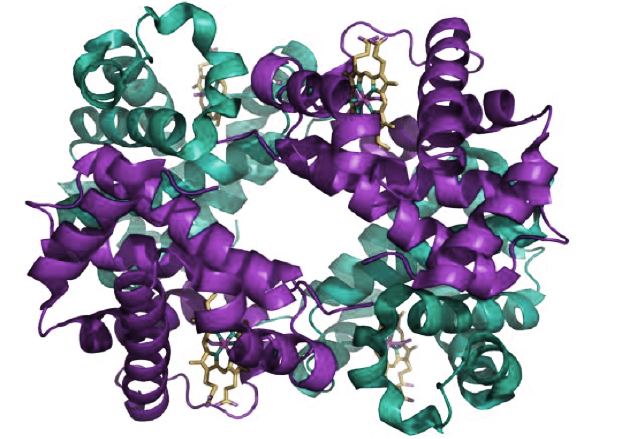
Estructura de la hemoglobina. Las subunidades proteicas se muestran en violeta y verde, y los grupos hemos, que contienen hierro, en amarillo. Fuente: Protein Data Base 1GZX
Hay proteínas muy cortas (en realidad se denominan péptidos), de unos pocos aminoácidos, y otras verdaderamente gigantes, como ciertas proteínas musculares, que llegan a tener hasta 100.000 aminoácidos.
Los ácidos nucleicos
Así como las proteínas están compuestas por aminoácidos, los ácidos nucleicos son polímeros de nucleótidos. Cada nucleótido está compuesto por una base nitrogenada, un fosfato y un azúcar. Hay dos tipos de ácidos nucleicos: el que tiene nucleótidos formados por el azúcar ribosa, es el ARN (ácido ribonucleico), y contiene las bases nitrogenadas A (adenina), G (guanina), C (citosina) y U (uracilo).
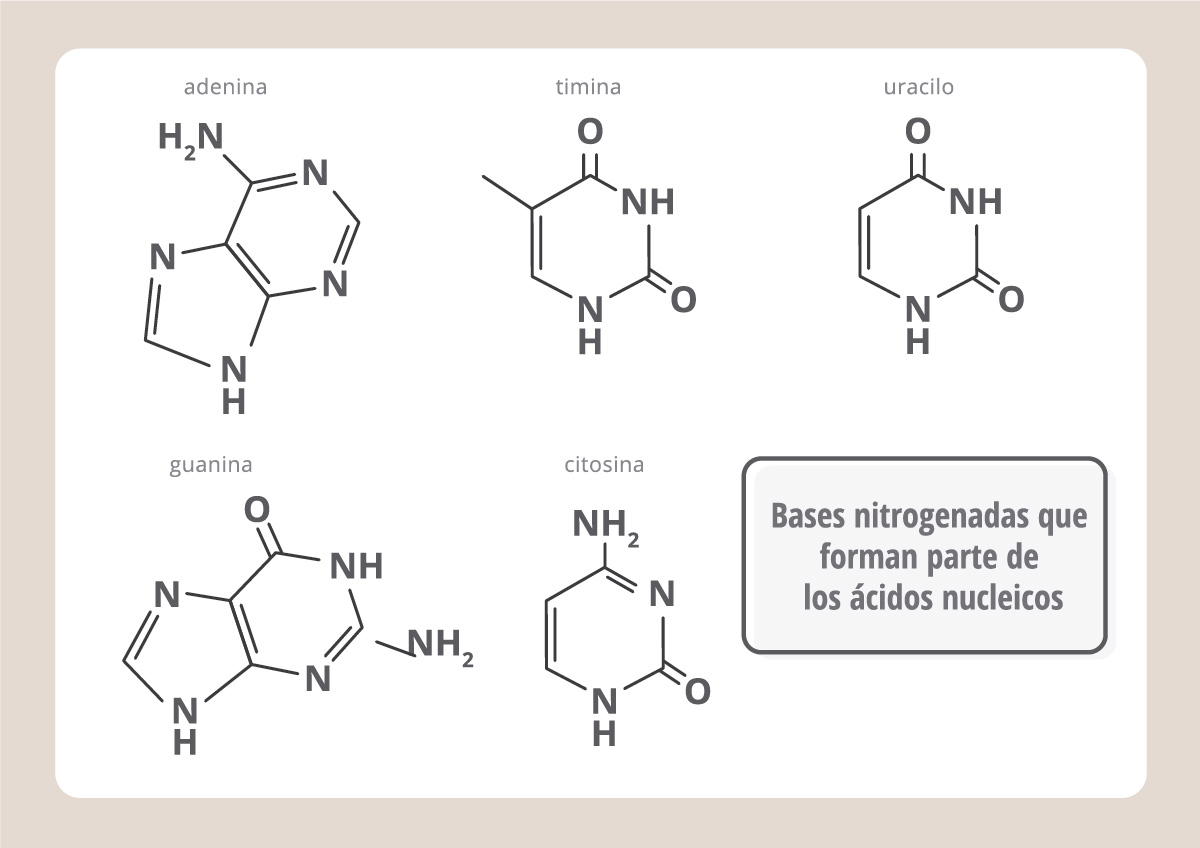
En cambio, el que tiene nucleótidos formados por el azúcar desoxirribosa es el ADN (ácido desoxirribonucleico) y contiene las bases nitrogenadas A (adenina), G (guanina), C (citosina) y T (timina, que es parecida a la U). Mientras que el ARN se encuentra en las células como una cadena polinucleotídica única, el ADN está formado por dos cadenas que se entrelazan formando una doble hélice.
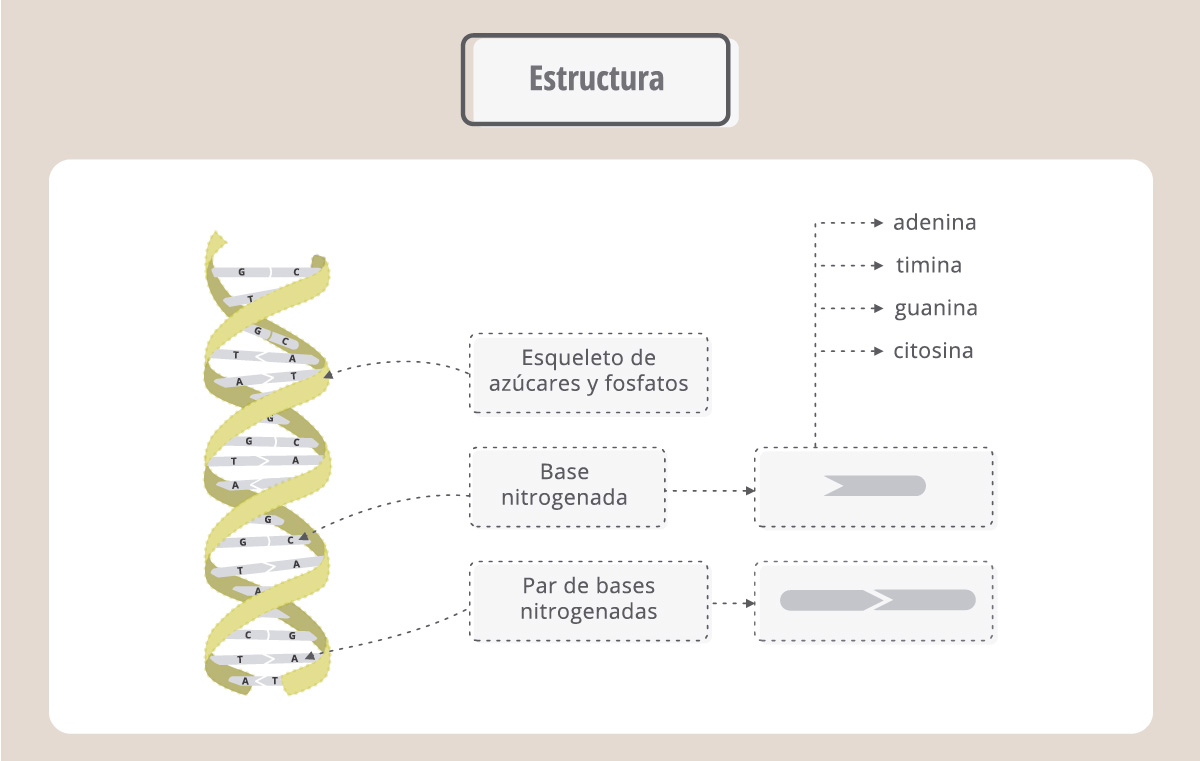
Podemos imaginar al ADN como una escalera que gira sobre sí misma y donde los lados son cadenas de azúcares y fosfatos, conectadas por “escalones”, que son las bases nitrogenadas. En la doble hélice, siempre una A se enfrenta a una T y una C se enfrenta a una G. Estas bases se unen entre sí a través de uniones no covalentes, conocidas como “puente de hidrógeno”.
- Cuaderno para docentes Nº 32: los ácidos nucleicos, estructura y función
2.4. El cultivo de células
Un cultivo celular es un proceso por el cual se mantienen y multiplican células procariontes o eucariontes, bajo condiciones controladas. De una manera más estricta, el término hace referencia al cultivo de células que derivan de organismos pluricelulares, como plantas y animales.
- Los cultivos celulares y sus aplicaciones I (cultivos de células animales)
- Los cultivos celulares y sus aplicaciones II (cultivos de células vegetales)
2.5. El ADN y los genes
¿Dónde están las instrucciones?
Las instrucciones que determinan todas las características y funciones celulares se encuentran en su material genético: el ADN.
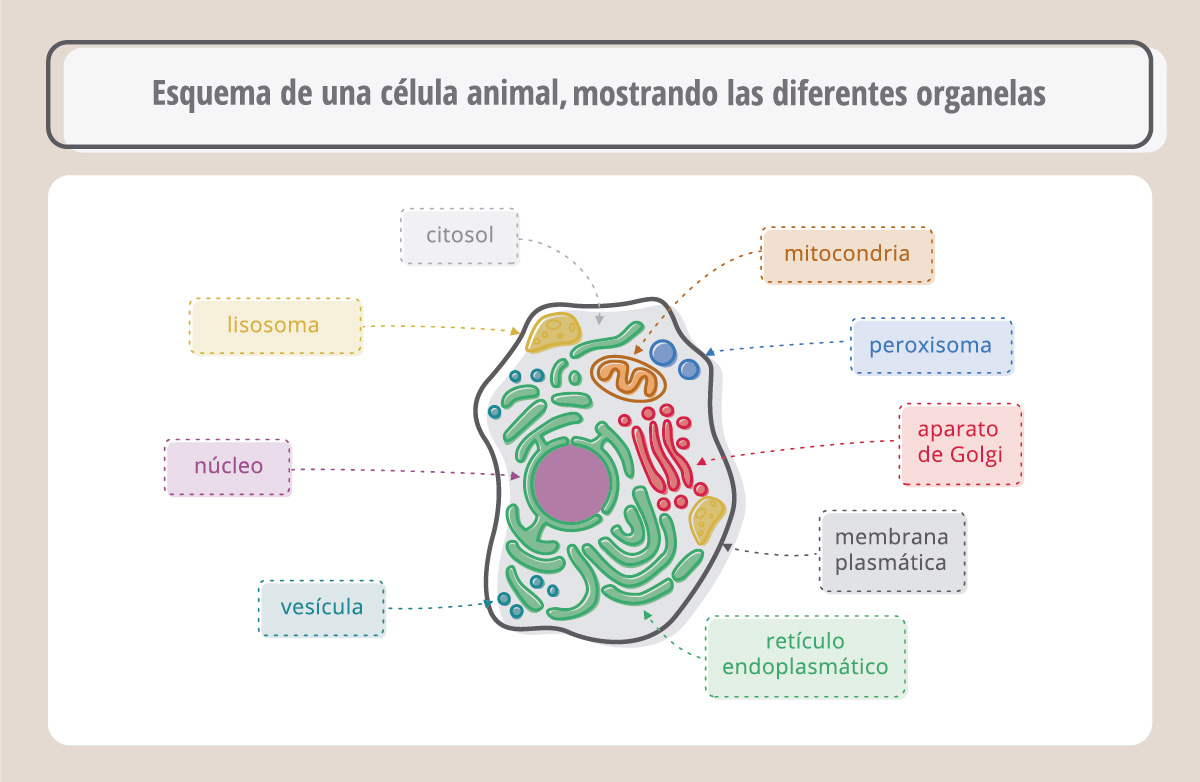
Veamos el esquema de una célula animal: está rodeada por una membrana plasmática y en su citoplasma se encuentran organelas que realizan diferentes funciones. Entre ellas, encontramos al núcleo, y es allí donde vamos a buscar las moléculas que queremos estudiar. En el núcleo encontramos a los cromosomas, que inmediatamente antes de la división celular adoptan una forma de X.
Fotografía de un cromosoma humano, obtenida por microscopía electrónica
Para cada especie, el número de cromosomas es fijo, los humanos tenemos 46 cromosomas por célula, agrupados en 23 pares, de los cuales 22 son autosomas y uno es sexual. (Una mujer tendrá un par de cromosomas sexuales XX y un varón tendrá un par XY).

Cada cromosoma tiene dos brazos idénticos, denominados cromátidas hermanas, unidas por una estructura llamada centrómero.
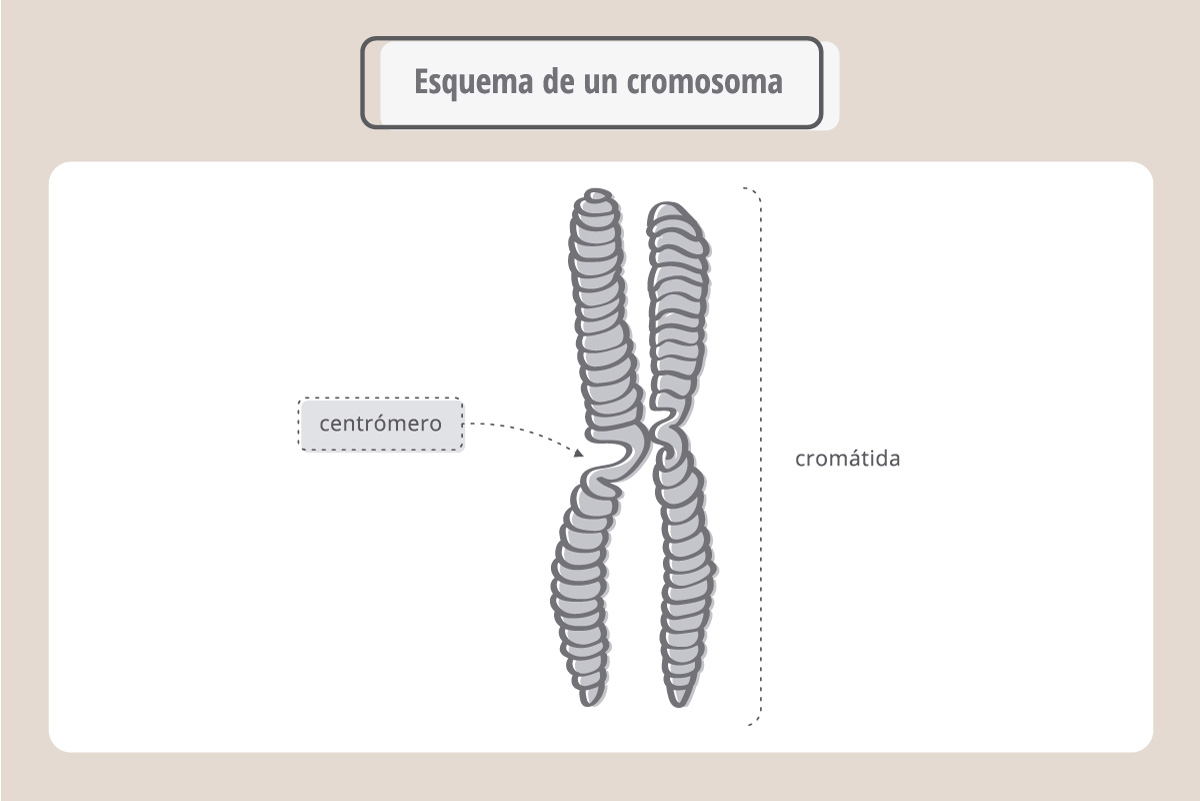
Estas cromátidas se van a separar a nivel del centrómero en el momento de la división celular. Este proceso, conocido como mitosis, es muy preciso, ya que asegura que cada célula hija reciba el mismo número de cromosomas y la misma información.

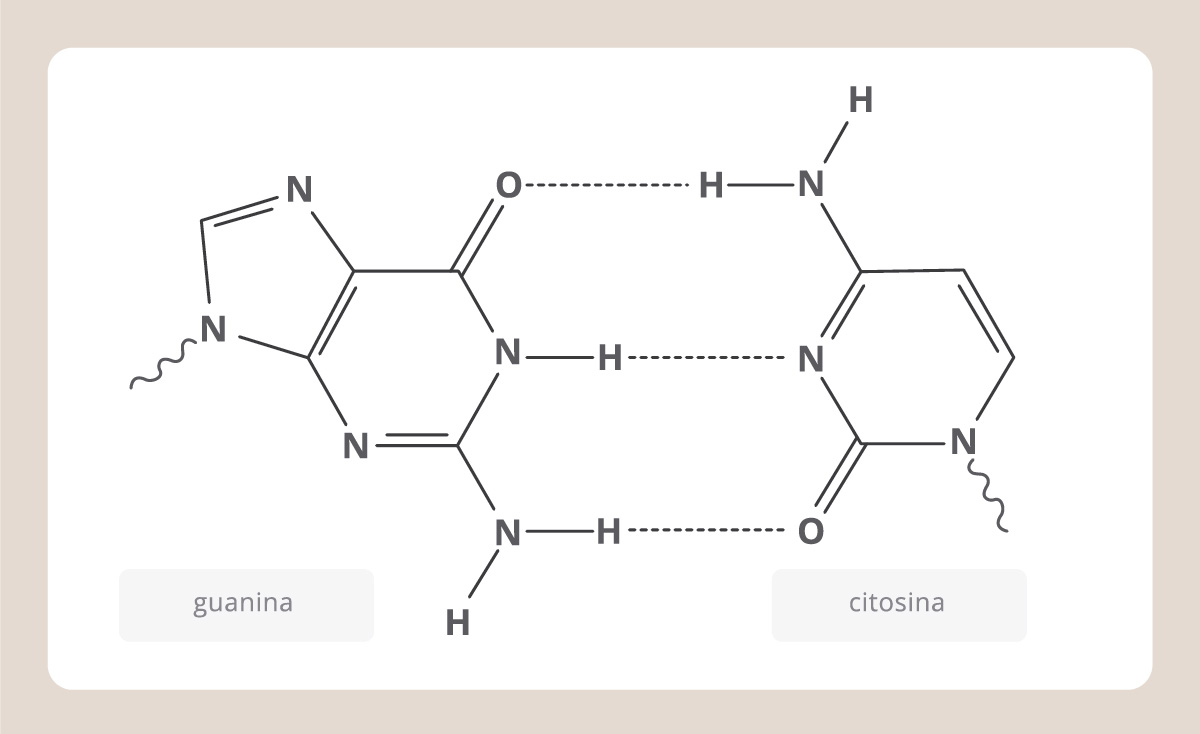
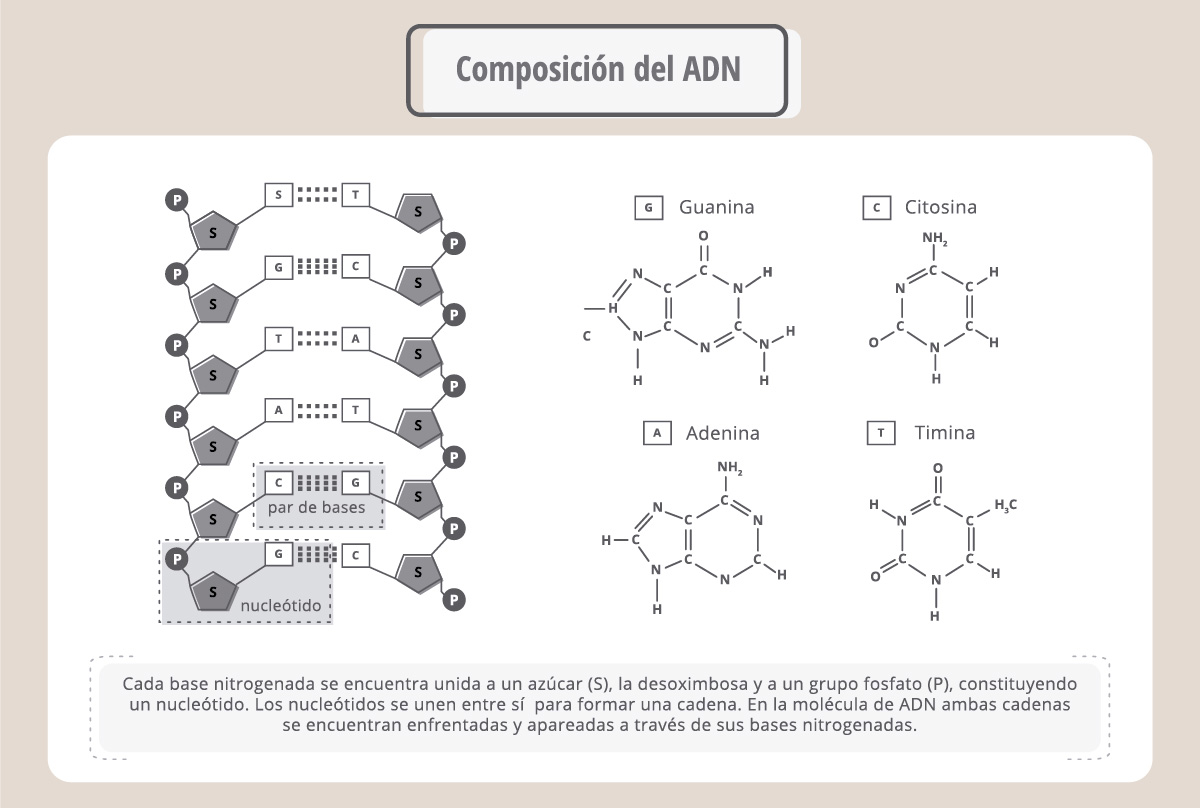
En esta figura vemos que cada cromátida del cromosoma está compuesta por una molécula muy enrollada: el ADN o ácido desoxirribonucleico. El ADN es la molécula que estábamos buscando. Se compone de dos cadenas, cada una formada por nucleótidos. Cada nucleótido está compuesto por un azúcar, la desoxirribosa, un fosfato y una base nitrogenada. Las bases nitrogenadas son cuatro: adenina (A), timina (T), citosina (C), y guanina (G), y, como vemos en el esquema, siempre una A se enfrenta a una T y una C se enfrenta a una G en la doble cadena.
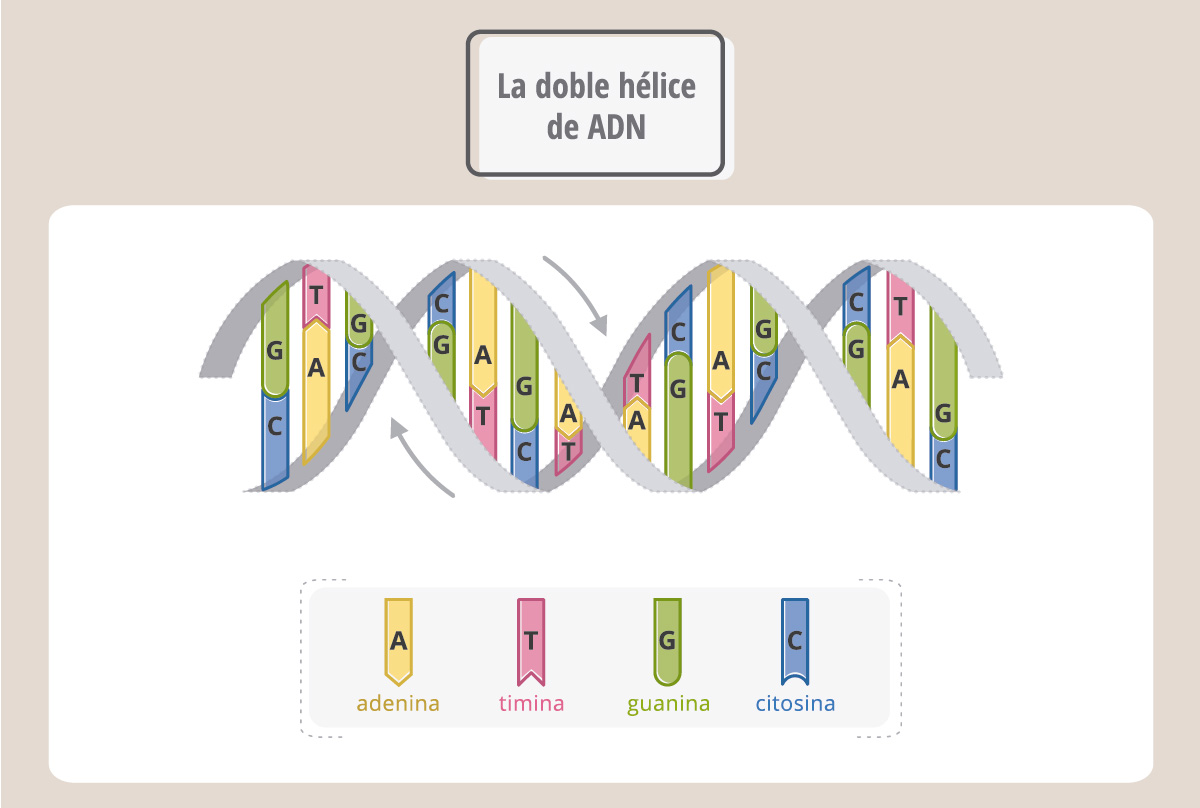
En el espacio, el ADN adopta una forma de doble hélice, denominada estructura de Watson y Crick, debido a los investigadores que la descubrieron. Podemos imaginar entonces al ADN como una escalera que gira sobre sí misma y donde los lados son cadenas de azúcares y fosfatos, conectadas por “escalones”, que son las bases nitrogenadas.
En realidad, la molécula de ADN se asocia a proteínas, llamadas histonas, y se encuentra muy enrollada y compactada para formar el cromosoma, que resulta ¡50.000 más corto que la molécula de ADN original!
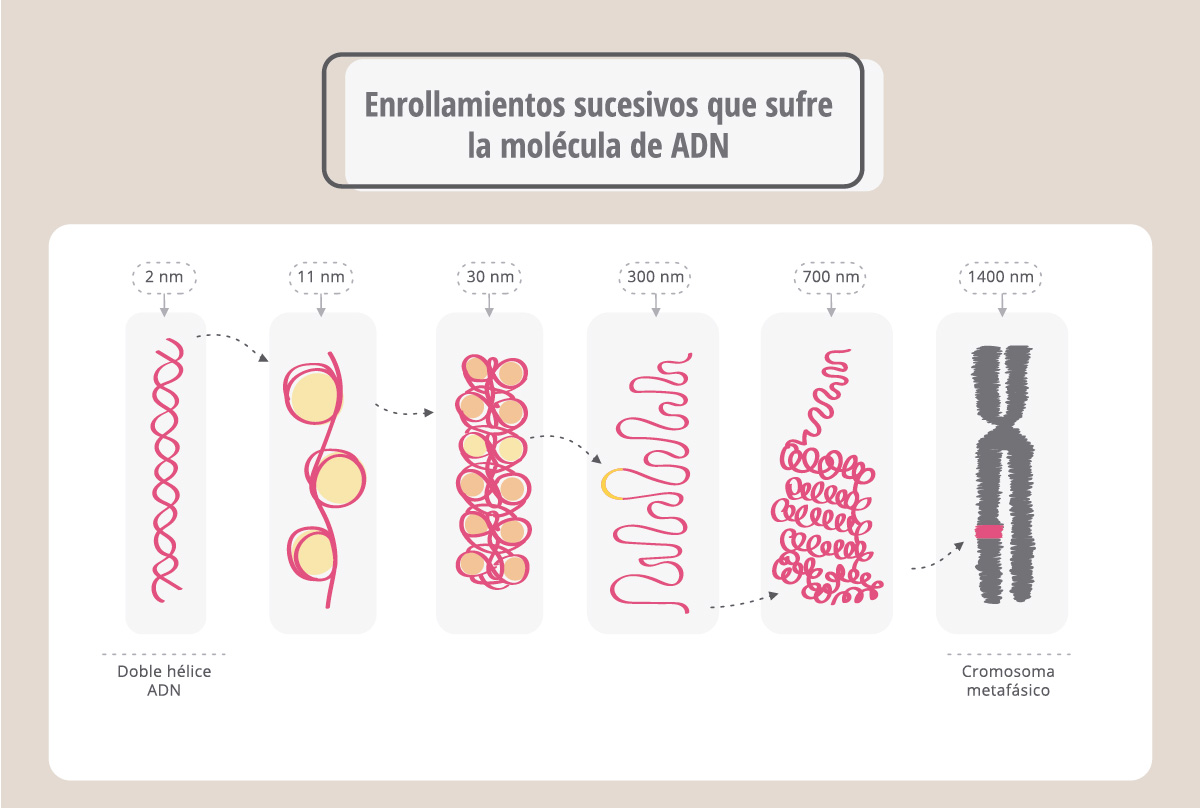
Hasta aquí podemos concluir entonces que un cromosoma está compuesto por dos cromátidas hermanas y que cada cromátida hermana está compuesta por una molécula de ADN. Durante la división celular las cromátidas hermanas se separarán, migrarán hacia los polos de la célula y finalmente constituirán los cromosomas de las células hijas. Cada célula hija iniciará más tarde su propia mitosis y para ello cada uno de sus cromosomas deberá estar constituido por dos cromátidas. ¿Cómo ocurre esta duplicación? La clave es que la molécula de ADN tiene la capacidad de replicarse, es decir, de generar moléculas hijas idénticas a la original. Durante la replicación, la molécula de ADN se desenrolla, separando sus cadenas. Cada una de éstas servirá como molde para la síntesis de nuevas hebras de ADN complementarias a la original. Para eso, la enzima ADN-polimerasa coloca nucleótidos siguiendo la regla de apareamiento A-T y C-G.
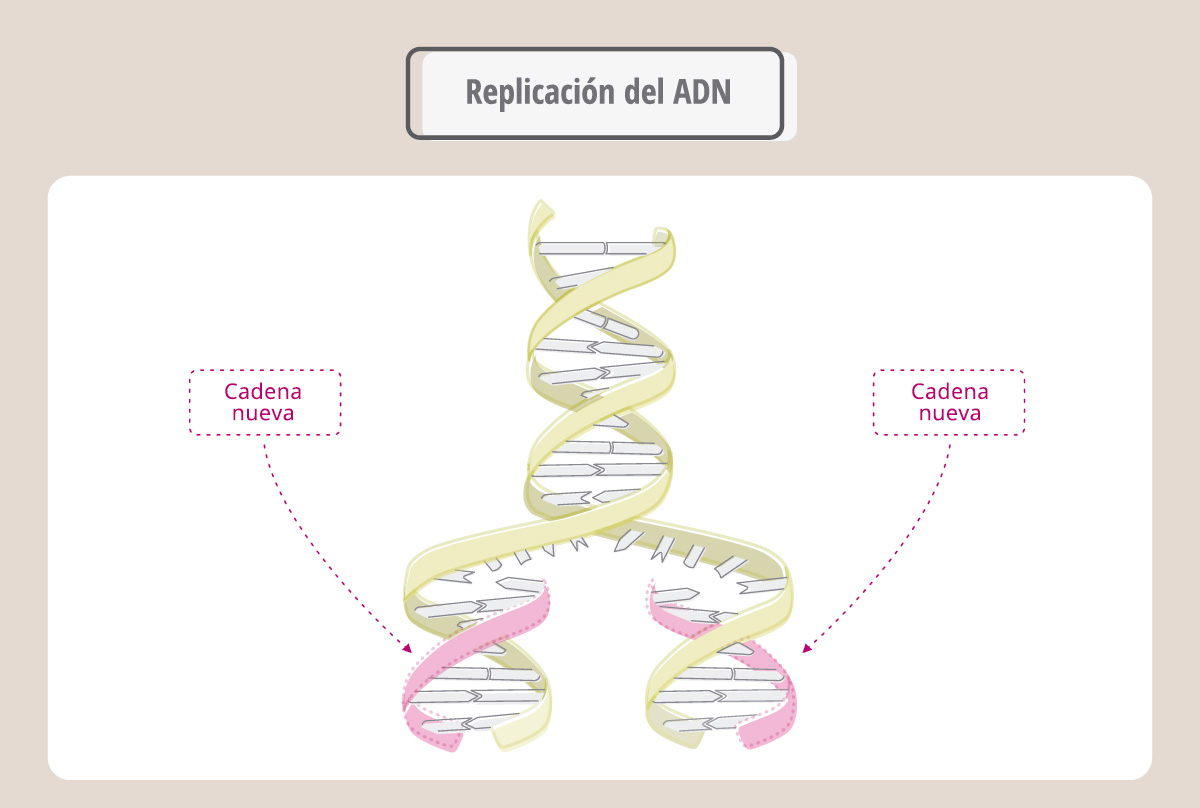
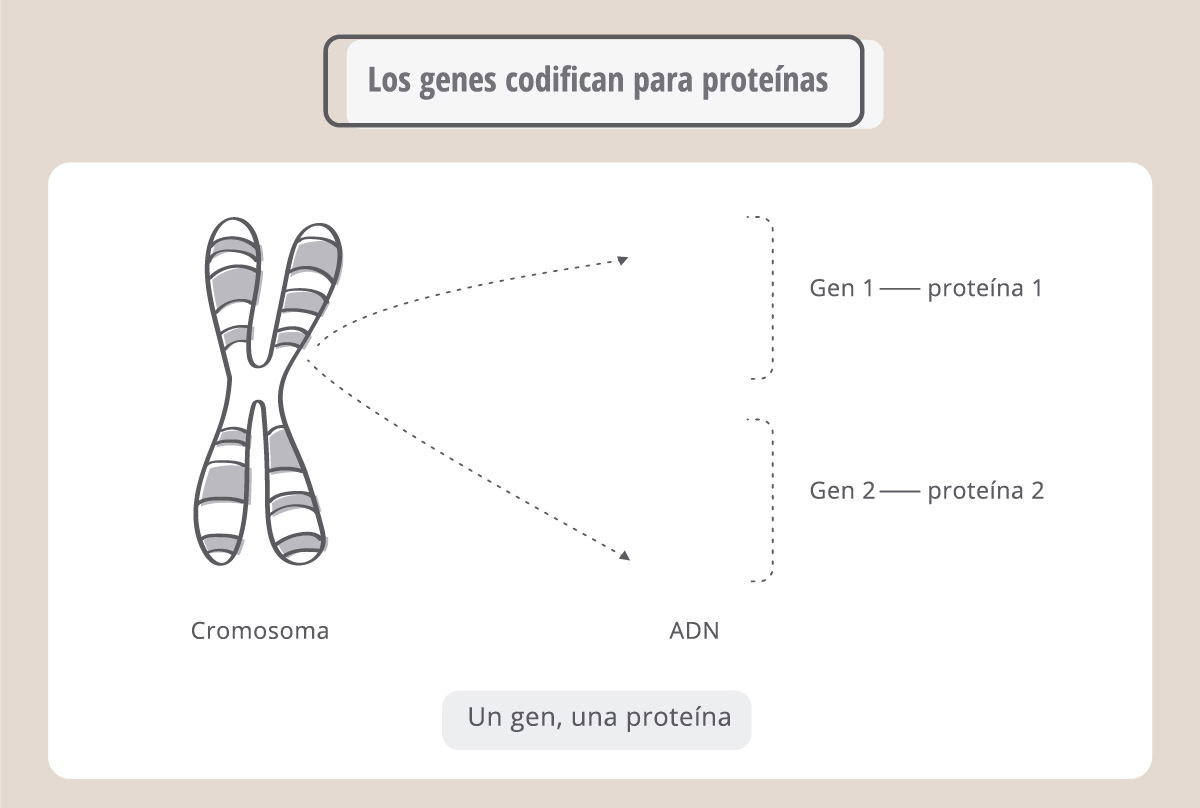
¿Cómo se interpretan las instrucciones?
La información guardada en el ADN es una secuencia de bases A, T, C y G que se combinan para originar “palabras” denominadas genes. Los genes son fragmentos de ADN cuya secuencia nucleotídica codifica para una proteína*.
Las proteínas son macromoléculas fundamentales para las funciones celulares (hay proteínas estructurales, otras son enzimas, otras transportan oxígeno, como la hemoglobina, y hay otras involucradas en la defensa inmunitaria, como los anticuerpos). Así como el ADN está compuesto por nucleótidos, las proteínas están compuestas por aminoácidos.
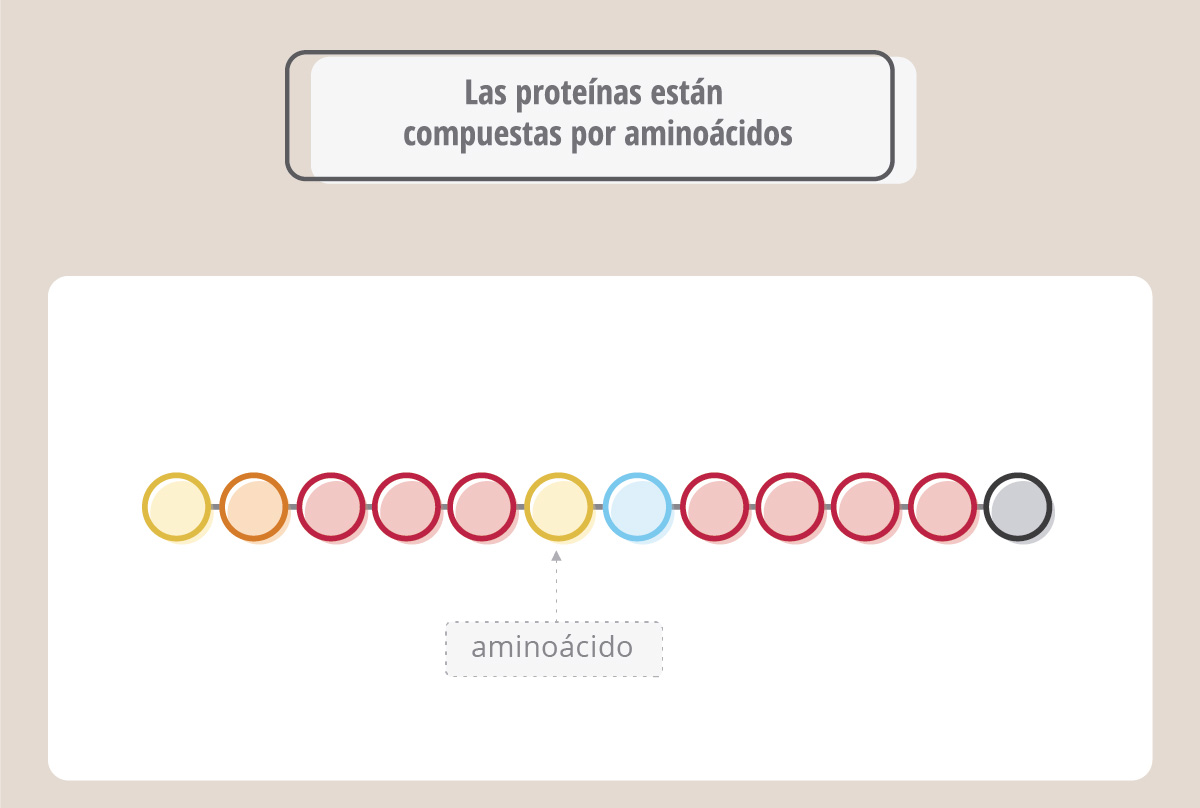

Hay 20 aminoácidos, y cada proteína tiene una secuencia de aminoácidos particular.
*Nota: En realidad, los genes son secuencias específicas de nucleótidos que llevan la información necesaria para la fabricación no sólo de proteínas sino también de moléculas de ARN, como los ribosomales y de transferencia.
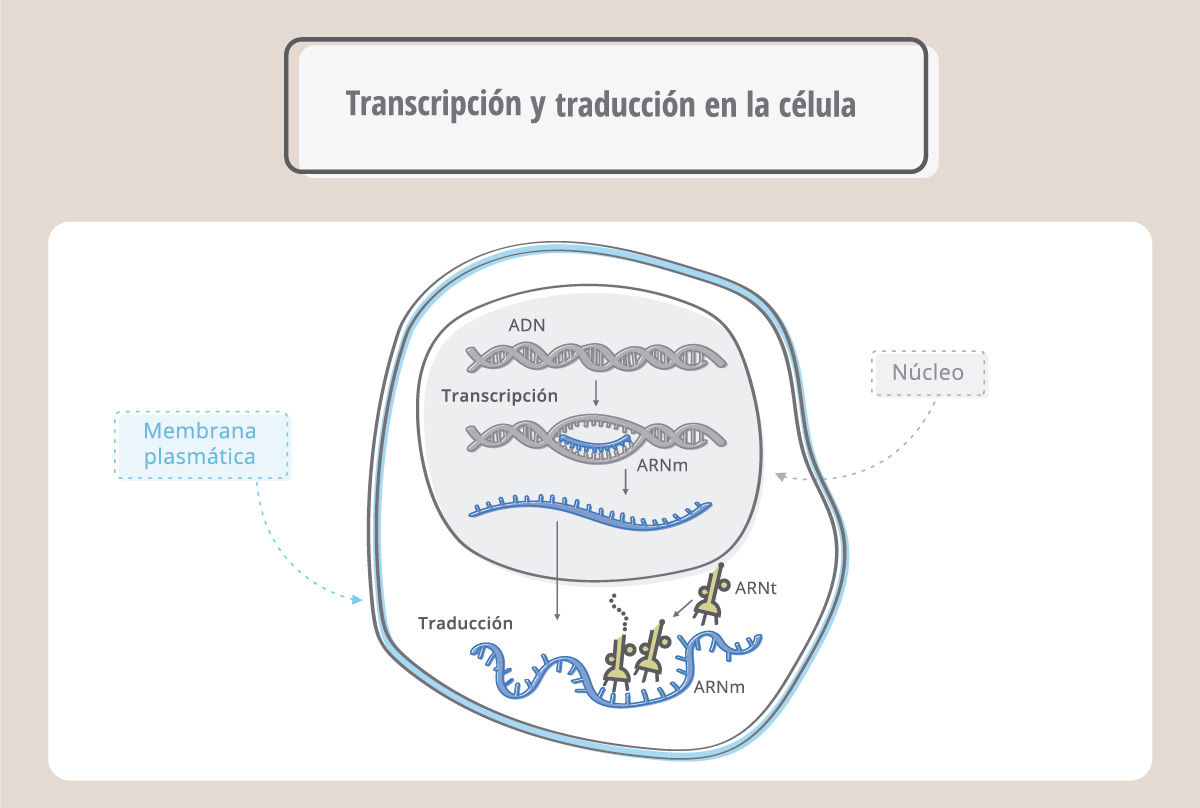
Las palabras (genes) escritas en el ADN en el lenguaje de los nucleótidos primero se copian o transcriben a otra molécula, el ARN mensajero, y luego se traducen al idioma de las proteínas, el de los aminoácidos. Este flujo de información se conoce como el “dogma central de la biología”.
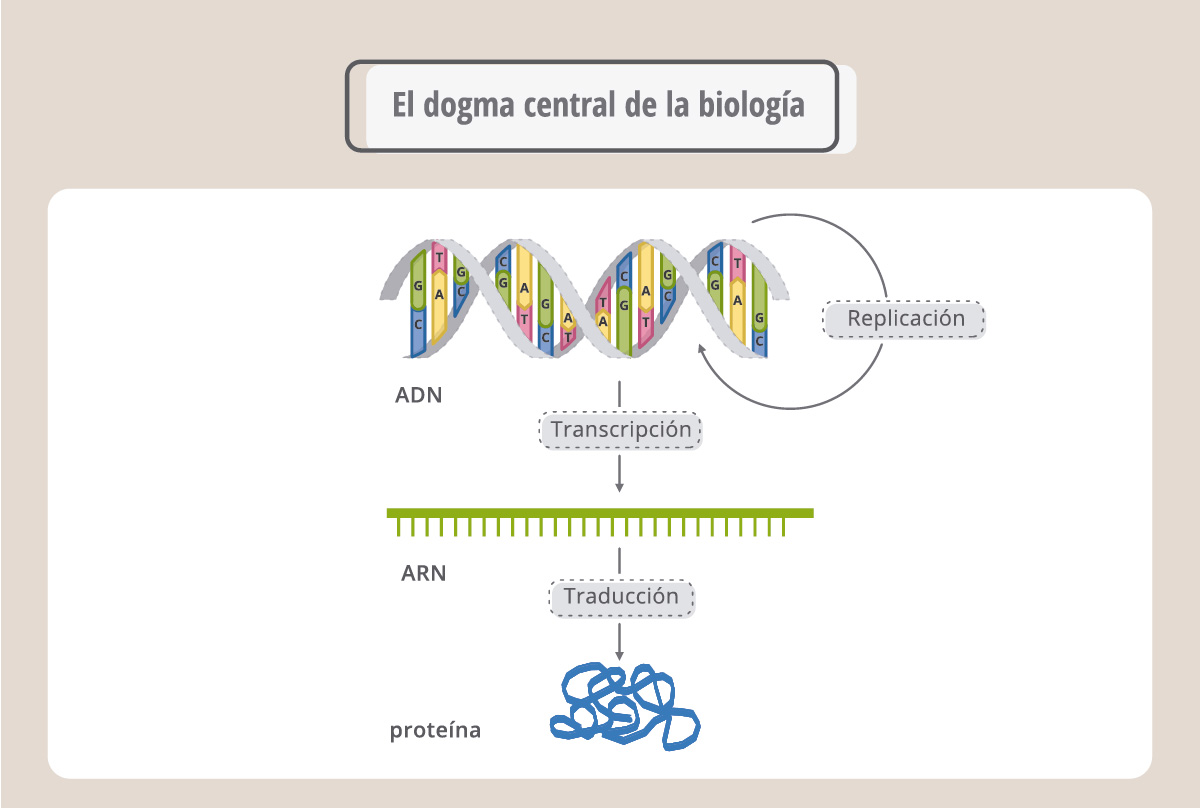
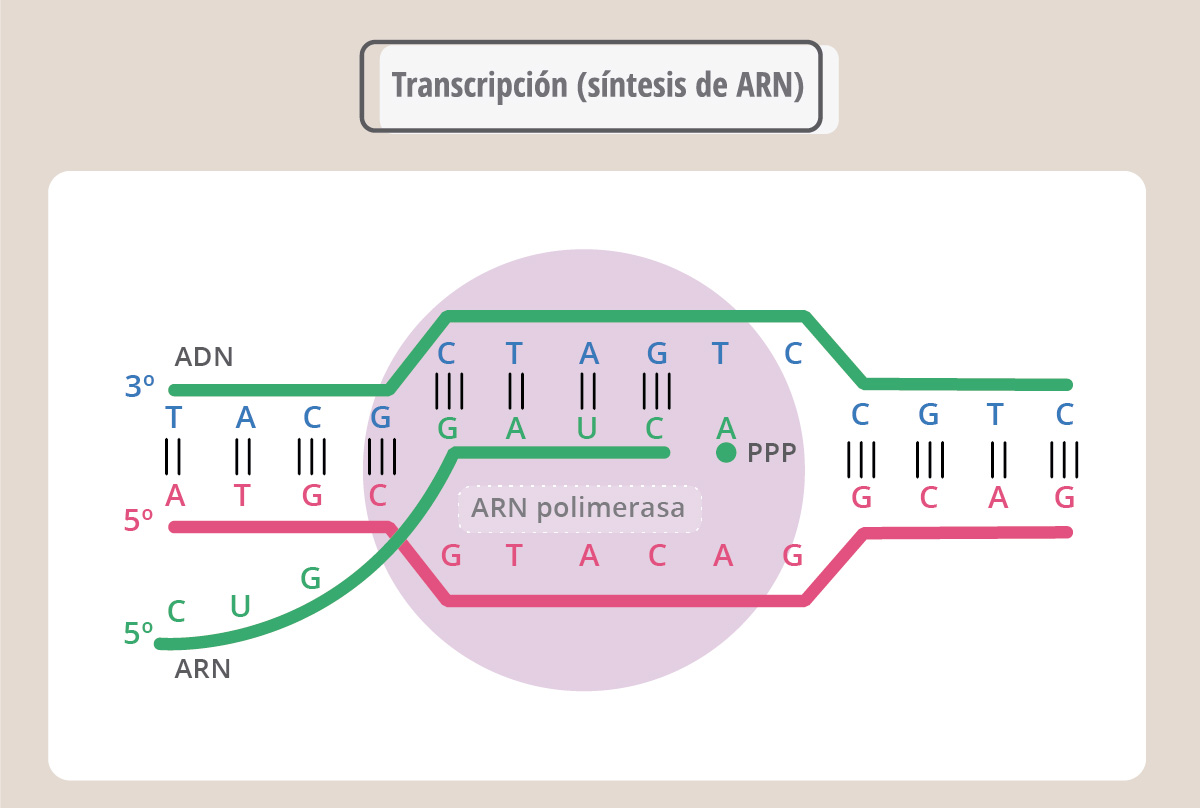
La transcripción es el proceso por el cual una enzima, denominada ARN polimerasa, copia la secuencia de ADN fabricando ahora ARN. El proceso es similar a la replicación, pero ahora la molécula nueva, de cadena simple, es ARN. Se denomina ARN mensajero porque va a llevar la información para que la maquinaria de síntesis proteica fabrique la proteína correspondiente. El ARN, o ácido ribonucleico, es similar al ADN aunque no igual. Se diferencia de éste en que es de cadena simple, en lugar del azúcar desoxirribosa tiene ribosa, y en lugar de la base nitrogenada timina, (T), tiene uracilo (U). Así, como muestra la esta figura, durante la síntesis del ARN, la enzima ARN polimerasa leerá en el ADN una A y colocará una U.
Para hacer una proteína, el ARN mensajero se lee cada tres nucleótidos (triplete). Cada triplete de bases se denomina codón.
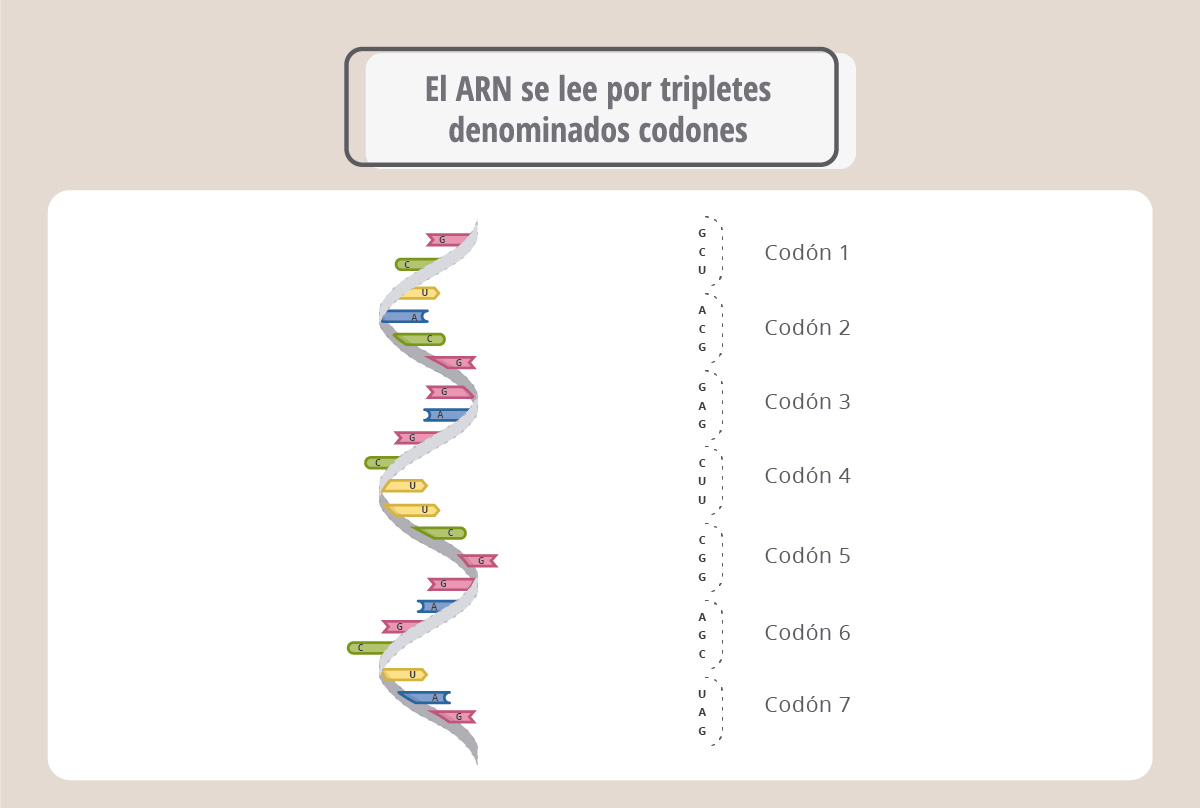
Si consideramos la combinación de cuatro bases tomadas de a tres, tenemos un total de 64 codones posibles. A cada codón le corresponde un aminoácido.
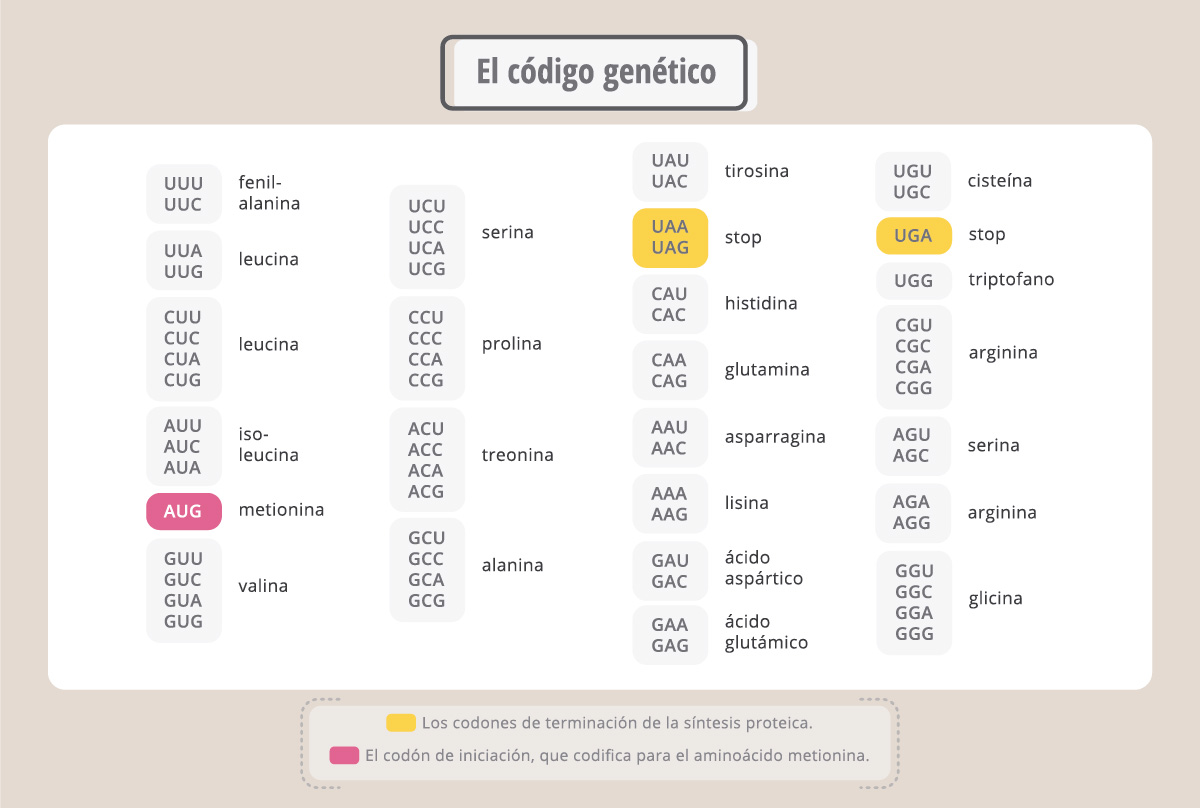
Esta tabla es el “diccionario” que nos permite traducir la información escrita en el lenguaje de los ácidos nucleicos (en nucleótidos) al lenguaje de las proteínas (aminoácidos), y es universal, o sea, es válido para todos los seres vivos. Así, la secuencia ATG (AUG en el ARNm) codifica para el aminoácido metionina, y el codón TTT (UUU en el ARNm) codifica para el aminoácido fenilalanina en todos los organismos vivos.
El código genético consiste en 61 codones que corresponden a aminoácidos y 3 codones de terminación (codones stop), responsables de la finalización de la síntesis proteica. Como sólo existen 20 aminoácidos en la naturaleza, varios codones pueden codificar para el mismo aminoácido (por ejemplo, al aminoácido glicina le corresponden los codones GGU, GGC, GGA y GGG).
El código genético fue elucidado por Marshall Nirenberg y Heinrich Matthaei, diez años después de que Watson y Crick describieran la estructura de doble hélice del ADN. Descubrieron que el ARN, independientemente del organismo del cual era aislado, podía iniciar la síntesis de proteínas cuando se lo incubaba junto a extractos celulares. Agregando un ARN sintético formado sólo por uracilos (poli-U), determinaron que el codón UUU (el único posible en el ARN poli-U) codificaba para el aminoácido fenilalanina, ya que el único producto que aparecía en el tubo era un polipéptido que contenía sólo este aminoácido. De la misma manera, un ARN artificial que consistía en nucléotidos A y C alternados originaba un polipéptido formado por histidinas y treoninas. Así, observando los productos formados luego de la incubación con una serie de ARN sintéticos, estos investigadores consiguieron descifrar el código genético.
Cada codón del ARNm en realidad es leído por otro ARN, llamado ARN de transferencia (ARNt), que actúa como un “adaptador” entre la información que lleva el ARNm y los aminoácidos que deben ir colocándose para formar la proteína correspondiente. El ARNt es muy pequeño comparado con los ARNm o ARN ribosomales, y tiene una estructura particular, denominada "hoja de trébol".
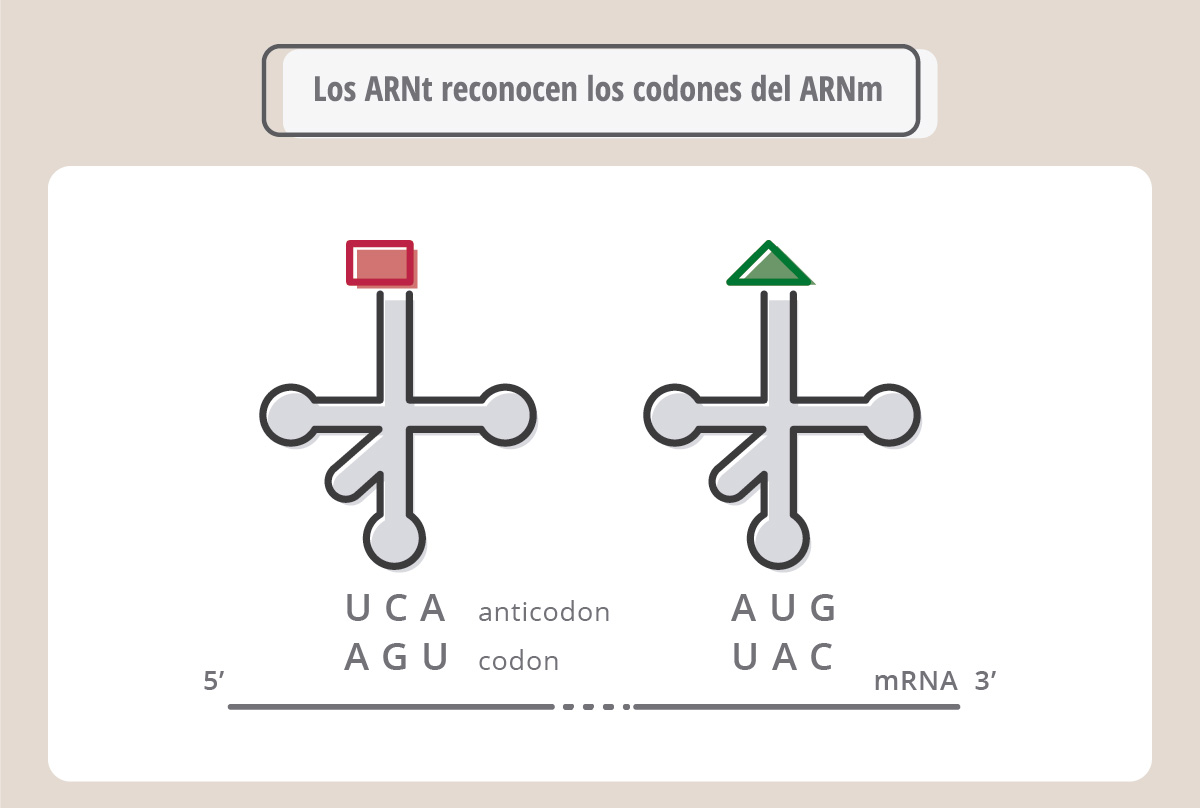
El ARNt tiene una secuencia, denominada anticodón que aparea (es decir, es complementaria) con el codón. Cada ARN de transferencia tiene un anticodón y “carga” un aminoácido en particular. Por ejemplo, como se muestra en la Fig. 18, el ARNt que tiene el anticodón UCA, se aparea al codón AGU, y carga el aminoácido serina (Ser). De la misma manera, el ARNt que carga tirosina (Tyr) se aparea, a través de su anticodón, con el codón UAC.
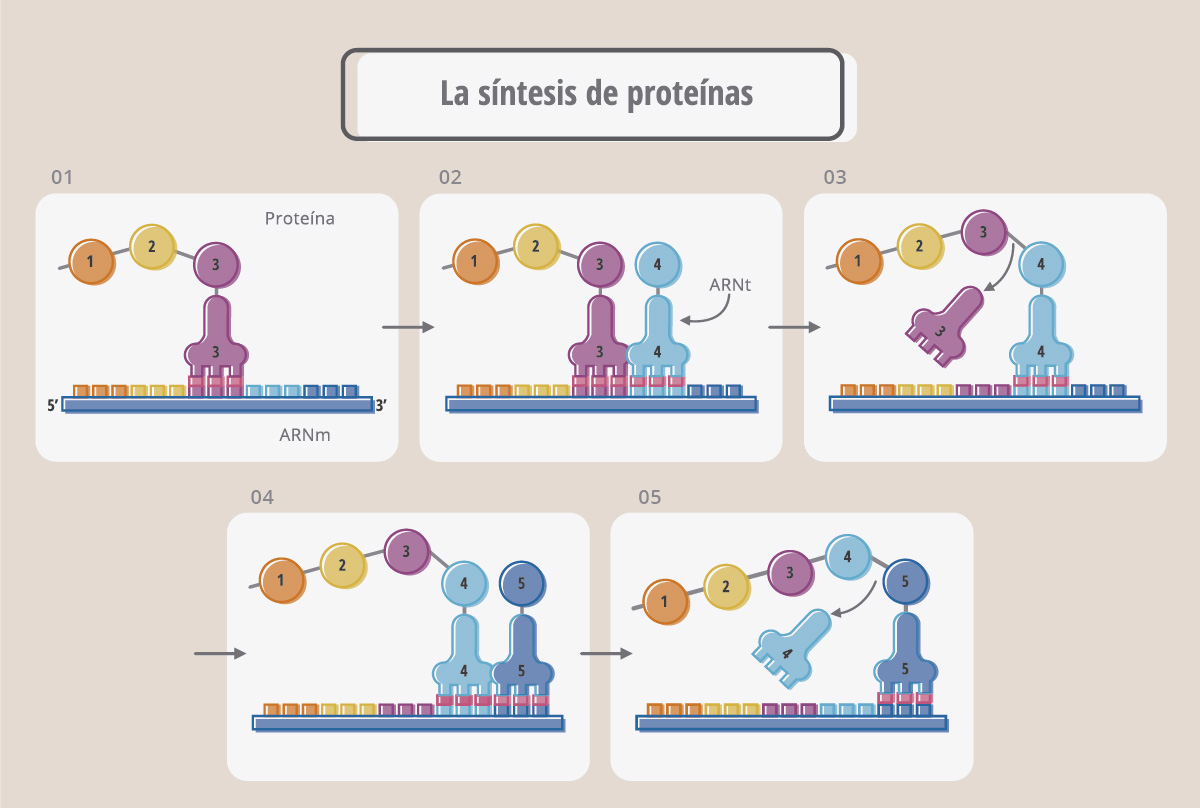
La figura muestra cómo se va formando una cadena polipeptídica (proteína) a medida que los ARNt reconocen sus respectivos codones. Este proceso complejo de síntesis proteica se denomina traducción y ocurre sobre los ribosomas.
Tanto la replicación del ADN como la transcripción ocurren en el núcleo. El ARNm recién sintetizado viaja luego al citoplasma donde se traduce para originar la proteína correspondiente.
No todos los genes se expresan al mismo tiempo
Todas nuestras células contienen dos juegos de 23 cromosomas (salvo los óvulos y espermatozoides que contienen sólo uno) y las “instrucciones” guardadas en cada juego de cromosomas de cada una de nuestras células son las mismas. Sin embargo, una neurona es una célula con prolongaciones y su función es recibir y transmitir impulsos nerviosos.
Neuronas
Glóbulo blanco
Los glóbulos blancos son en cambio redondeados y se ocupan de la defensa de nuestro cuerpo, reconociendo a los microorganismos patógenos para eliminarlos... ¿Si todas las células tienen las mismas “instrucciones” por qué no son todas iguales, tienen la misma forma e idéntica función?
Si bien todas las células de nuestro cuerpo tienen los mismos genes, no todos se expresan en todas las células, es decir, no todos se transcriben y traducen.
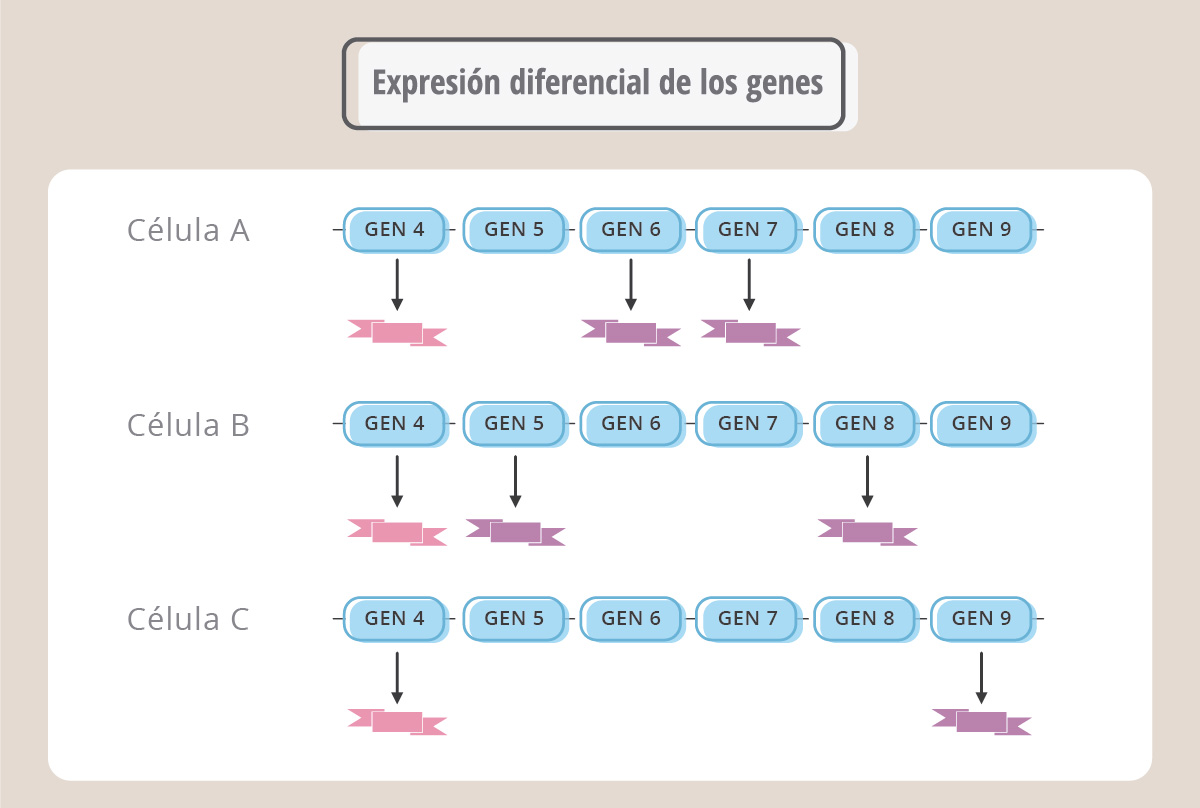
Por ej., el gen 4 se expresa en todas las células, mientras que en la célula A se producen específicamente las proteínas correspondientes a los genes 6 y 7, mientras que las proteínas derivadas de los genes 5 y 8 son exclusivas de la célula B y el producto del gen 9 es exclusivo de la célula C. En este ejemplo hipotético, el producto del gen 4 cumpliría funciones comunes a los tres tipos celulares mientras que la expresión diferencial de los otros genes determinaría las diferentes formas y funciones.
A veces los genes cambian (mutaciones)
A veces, y este es un fenómeno relativamente frecuente, la enzima que se encarga de la replicación (ADN polimerasa) se equivoca, es decir, coloca un nucleótido en lugar de otro. Veamos qué puede ocurrir entonces.
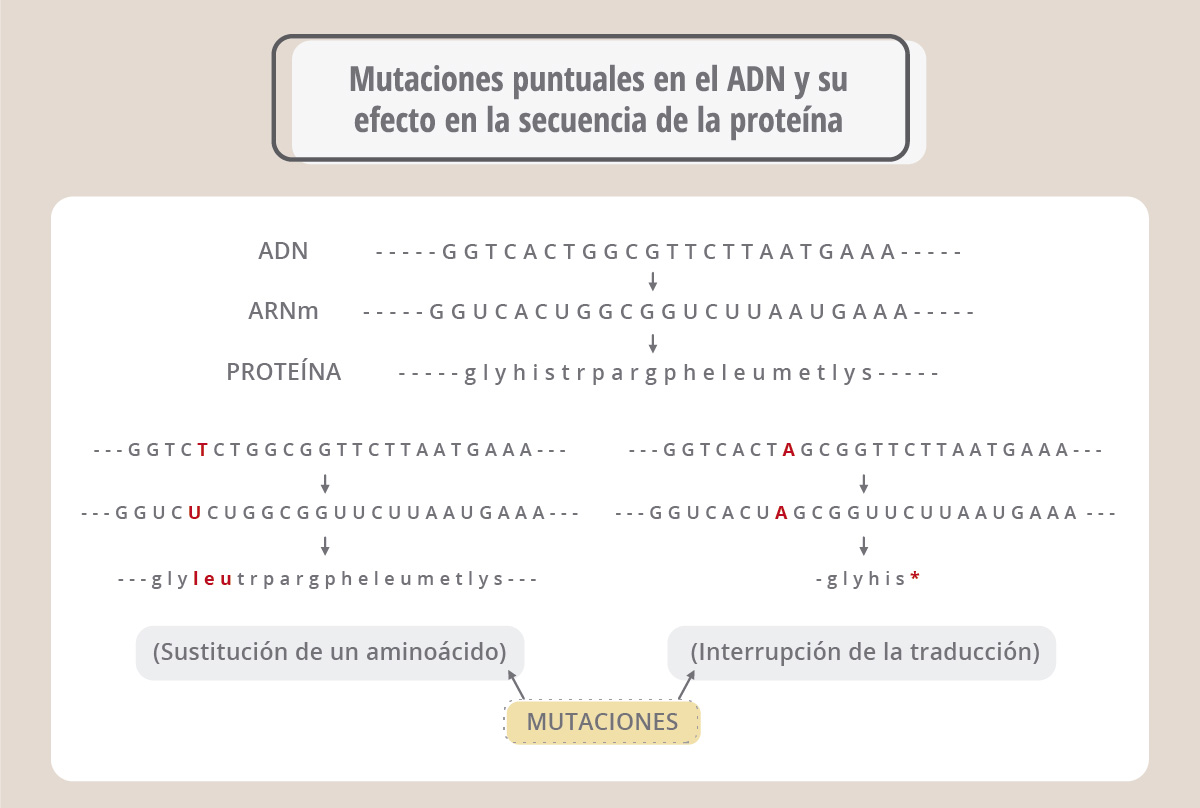
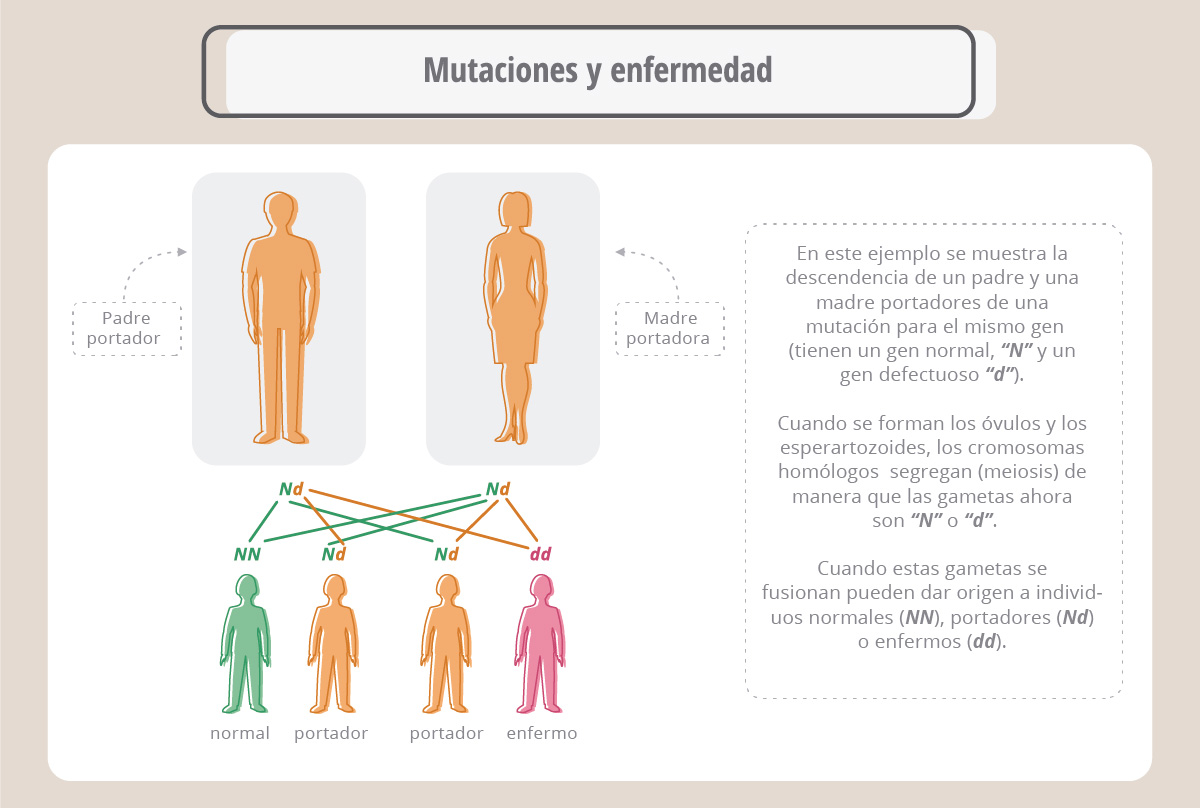
En el panel superior (ADN) vemos la secuencia de un fragmento de un gen; cuando éste se transcribe origina un ARN mensajero (ARNm), el cual es leído por la maquinaria de traducción para dar una proteína cuya secuencia de aminoácidos se ve más abajo (proteína). Supongamos ahora (panel de la izquierda) que la enzima ADN polimerasa se equivocó y colocó una T en lugar de la quinta A de la secuencia. Al traducirse el mensajero correspondiente, aparece una leucina en lugar de una histidina, porque el codón CAC original ahora es CUC. Por lo tanto, la proteína generada es diferente en un aminoácido a la original. Este cambio podría alterar o anular la función de la proteína. En el panel de la derecha vemos otro ejemplo, donde el codón UGG cambia a UAG, pero UAG es un codón de “parada”, o sea que la nueva proteína ahora es más corta, y probablemente no pueda realizar su función. Estos ejemplos ilustran el efecto de los cambios o mutaciones puntuales (debidos a un único cambio en la secuencia) en la proteína final. En algunos casos estas mutaciones pueden provocar la falta de actividad de una proteína esencial y causar una enfermedad. Las enfermedades genéticas surgen de mutaciones espontáneas que ocurren en las células germinales (óvulos y espermatozoides) y que por lo tanto se transmiten a las generaciones siguientes. Es importante recordar que cada cromosoma tiene su par homólogo, de manera que podemos ser portadores de una mutación sin que se vean afectadas nuestras funciones, ya que en el otro cromosoma homólogo el gen puede ser normal.
Un ejemplo de enfermedad debida a una mutación puntual es la anemia falciforme, causada por un cambio en la secuencia del gen de la b-globina, componente de la hemoglobina. En los pacientes que padecen esta enfermedad, los glóbulos rojos son más rígidos que lo normal y no pueden transitar fácilmente por los capilares sanguíneos. Otro ejemplo lo constituyen los diferentes tipos de hemofilia, causada por mutaciones puntuales en los genes que codifican para los factores de coagulación. Al no poseer todos los factores necesarios para la coagulación de la sangre, el paciente sufre hemorragias.
¿Las mutaciones siempre están asociadas a enfermedades? Absolutamente no. La mayoría de las mutaciones no se manifiestan, o porque están en regiones del ADN donde no hay genes, o porque no cambian el aminoácido (recordar que el código genético es degenerado), o porque ese cambio no altera la función de la proteína. O bien podría alterarse la función y esto no resultar perjudicial. Tal es el caso del carácter color de ojos, donde el color claro se produce por falta de ciertas enzimas que fabrican los pigmentos del iris. ¡Es obvio que tener ojos claros no significa estar enfermo! En realidad, las mutaciones son la base de la biodiversidad: todos los humanos tenemos el mismo genoma, pero con algunas poquísimas variaciones. Estas variaciones determinan que seamos diferentes en el color de ojos, en la altura, en la ondulación de nuestro cabello, etc. Sobre la biodiversidad actúa la selección natural... pero ese es otro capítulo.
2.6. El ADN como material genético (un poco de historia)
Los neumococos de Griffith
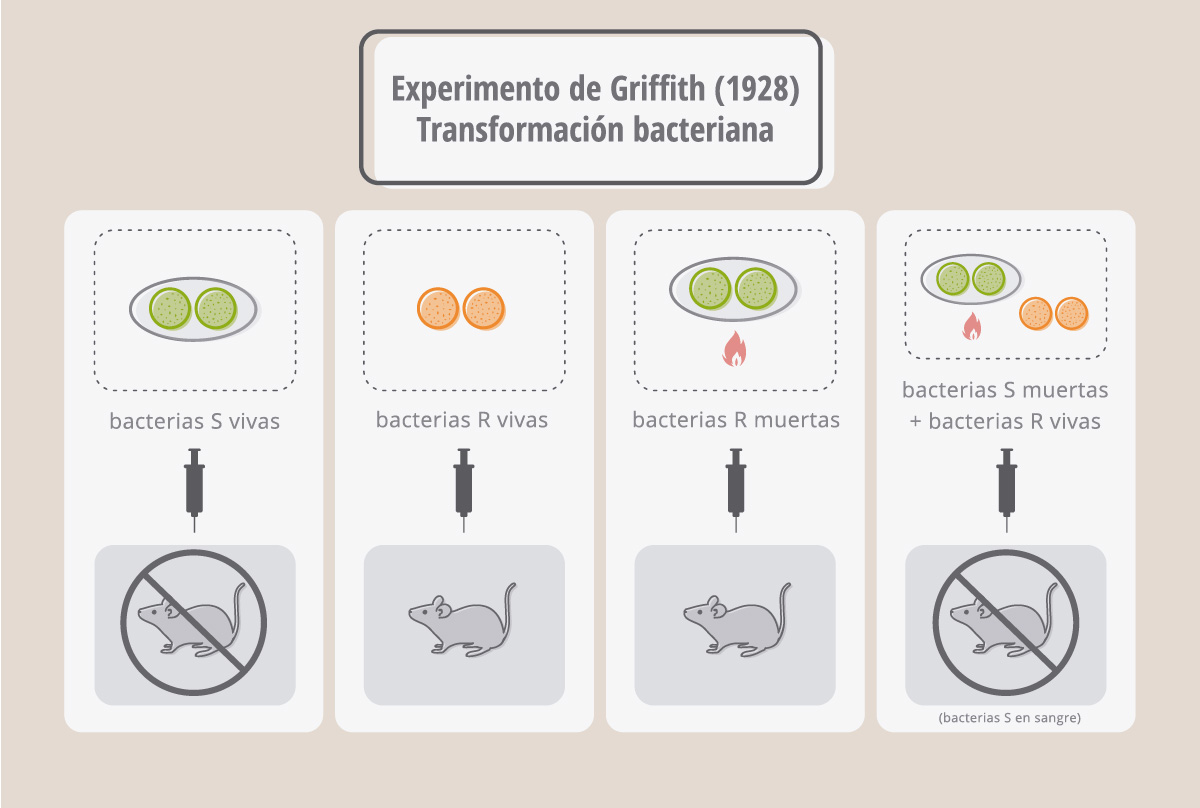
La historia del descubrimiento de la composición química de los genes se inicia en 1928, cuando el médico inglés Frederick Griffith realizaba sus experimentos de infección de ratones con los neumococos (bacterias que causan la neumonía en humanos). La inoculación de estas bacterias en los ratones causa su muerte en 24hs y su patogenicidad se debe a la cápsula de polisacáridos que poseen por fuera de su pared celular. Esta cápsula les otorga a las colonias de neumococos un aspecto brillante o liso, denominado S. Existen mutantes de neumococos que no producen la cápsula de polisacáridos y forman colonias de aspecto rugoso o R. Griffith descubrió que estas mutantes no mataban a los ratones. Pero, sin embargo, si mezclaba a los neumococos R con neumococos S previamente muertos por calor, entonces los ratones se morían. Aún más, en la sangre de estos ratones muertos Griffith encontró neumococos con cápsula (S). Esto quiere decir que en las bacterias S muertas había “algo” capaz de transformar a las bacterias R en patógenas, y este cambio era permanente y heredable! Más tarde se demostró que esta transformación también se producía si se incubaban los neumococos R con un extracto libre de células S.
¿Qué sustancia transmitía la propiedad de matar a los ratones de las bacterias S muertas a las bacterias R vivas? Esta pregunta es clave si consideramos a la transformación de R en S como un fenómeno de intercambio de información genética. Pero en ese entonces nadie imaginaba que las bacterias llevaran genes y por lo tanto la identificación de la sustancia responsable de tal transformación parecía no estar relacionada con el descubrimiento de la naturaleza de los mismos.
La naturaleza del principio transformante
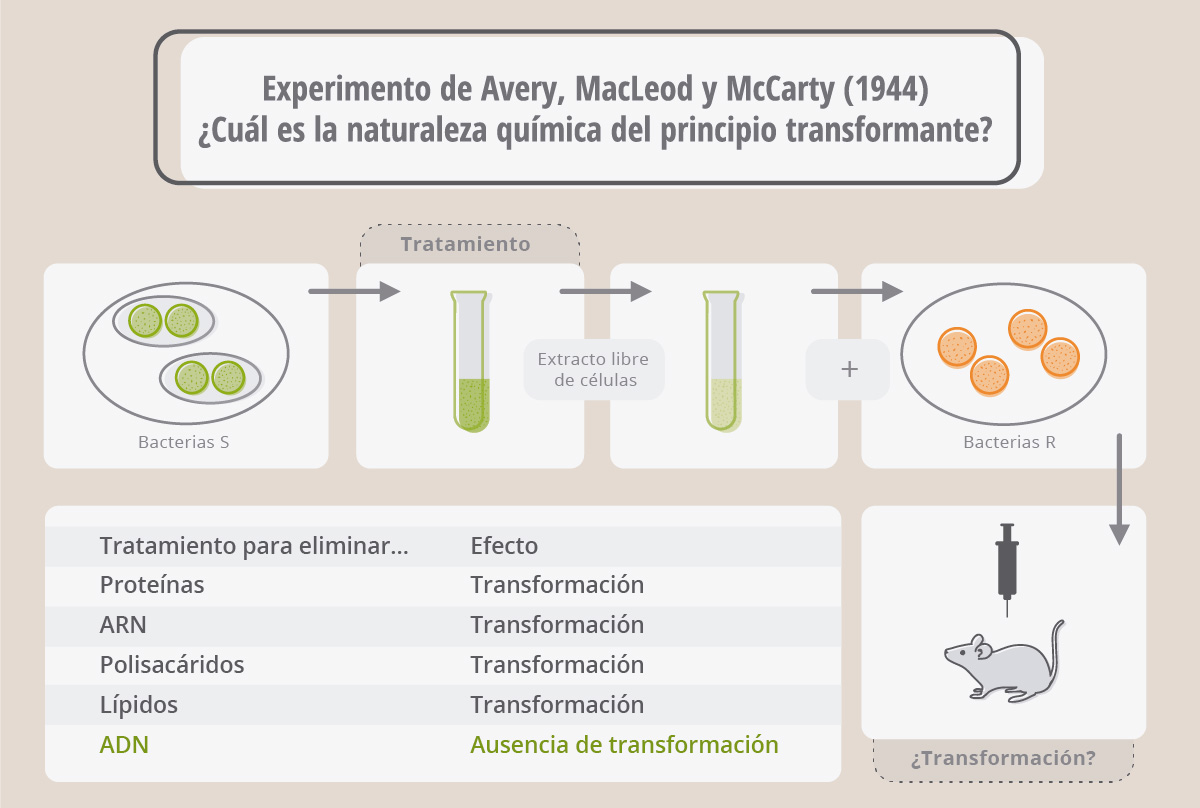
El médico microbiólogo Oswald Avery quedó sorprendido por los resultados publicados por Griffith y aunque al principio no creía mucho en ellos, se propuso descubrir la sustancia responsable del fenómeno de transformación. Así fue como Oswald Avery, junto a sus colegas Colin MacLeod y Maclyn McCarty comenzaron a fraccionar el extracto de bacterias S libre de células donde, según Griffith, estaba el principio transformante. Encontraron que podían eliminar las proteínas, los lípidos, los polisacáridos y el ARN del extracto sin disminuir la propiedad del extracto de transformar a los neumococos R en S. Sin embargo, si purificaban el ADN presente en el extracto y lo incubaban con las bacterias R, éstas se transformaban en S. Era el ADN el principio transformante que hacía que los neumococos R se transformaran en S, es decir, era el ADN el que llevaba la información necesaria para que la cepa R fuera capaz de sintetizar una cápsula de polisacáridos idéntica a la que poseían las bacterias S.
Cuando Avery, MacLeod y McCarty publicaron sus resultados en 1944, fueron muy pocos los que concluyeron que los genes estaban compuestos de ADN. En esa época era realmente difícil de imaginar que una molécula “monótona” compuesta sólo de cuatro bases nitrogenadas diferentes pudiera tener la suficiente variabilidad como para llevar toda la información genética que precisaban los seres vivos. Sin duda, eran las proteínas las candidatas para tal función, debido a su gran complejidad y múltiples formas. En este contexto, Avery, MacLeod y McCarty concluyeron, tímidamente, en su artículo:
“Si los resultados del presente estudio se confirman, entonces el ADN debe ser considerado como una molécula que posee especificidad biológica cuya base química aún no ha sido determinada”.
Los bacteriófagos de Hershey y Chase
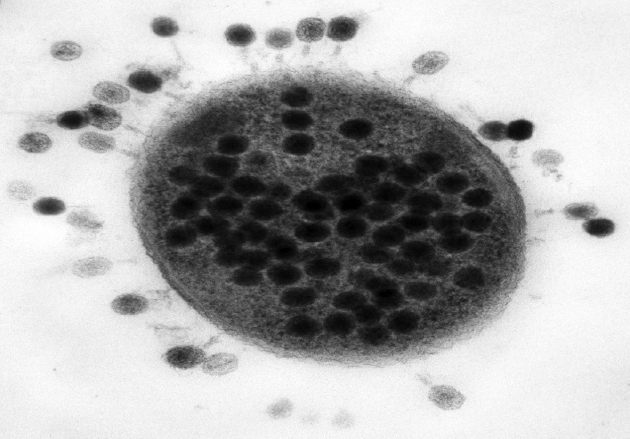
Llevó ocho años más para que la comunidad científica se convenciera de que el ADN era el material genético. Fue gracias al experimento que presentaron Al Hershey y Martha Chase en 1952, sobre la infección de bacteriófagos o fagos (virus que infectan bacterias). Los fagos están compuestos por una cabeza proteica que guarda en su interior ADN. Hershey y Chase vieron que durante la infección el ADN abandona la cabeza del fago y entra en la bacteria, dejando afuera la cabeza proteica. Es decir que el ADN lleva la información necesaria y suficiente para hacer más fagos hijos dentro de la bacteria. En otras palabras, el experimento indicaba que era el ADN el portador de la información genética del fago.
La conclusión de que el ADN portara la información genética para la continuidad de los fagos coincidía plenamente con la obtenida por Avery, MacLeod y McCarty, que indicaba que el ADN era el material genético de las bacterias. Sin embargo, y después de la desconfianza con que habían sido tomados los resultados sobre la transformación bacteriana, fue el experimento de los fagos el que disipó las dudas sobre la composición química de los genes.
El ADN no es tan aburrido como parece
Como mencionamos anteriormente, para esa época prevalecía la idea de que el ADN era una molécula demasiado “aburrida” como para ser considerada portadora de la información genética. Esta idea fue desechada gracias al trabajo de Erwin Chargaff, quien analizó en detalle la composición de bases del ADN extraído de diferentes organismos. Llegó a la sorprendente conclusión de que las bases nitrogenadas no se encontraban en proporciones exactamente iguales en levaduras, bacterias, cerdos, cabras y humanos, sugiriendo que el ADN no debía ser tan monótono. Sin embargo, demostró que, independientemente del origen del ADN, la proporción de purinas era igual a la de pirimidinas, y que la proporción de adeninas era igual a la de timinas, y la de citosinas igual a la de guaninas. En su artículo, publicado en 1950, señaló:
“Los resultados ayudan a refutar la hipótesis del tetranucleótido. Es sin embargo notable, aunque no podemos decir que este hallazgo no sea más que accidental, que en todos los ADN examinados las proporciones entre el total de purinas y el total de pirimidinas, así como entre adenina y timina, y citosina y guanina, fueron próximos a 1”
Este resultado reflejaba por primera vez un aspecto estructural del ADN, indicaba que independientemente de la composición de A o de G en un ADN, siempre la concentración de A es igual a la de T y la de C igual a la de G. Sin embargo, en aquel momento Chargaff no sospechó las implicancias que podían tener estas reglas (denominadas más tarde “reglas de Chargaff”) en la elucidación de la estructura del ADN. Ni siquiera queda claro si Watson y Crick las tuvieron en cuenta para postular el modelo de la doble hélice.
Finalmente, la doble hélice
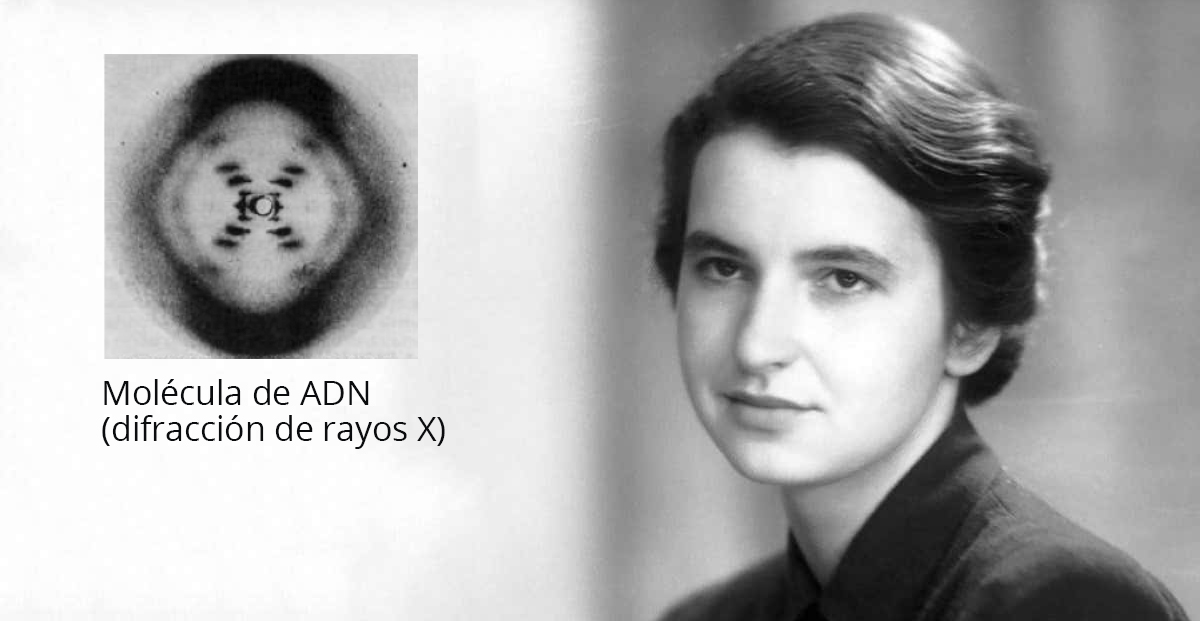
A comienzos de la década de 1950, tres grupos de investigadores trabajaban simultáneamente en la estructura del ADN. Uno de ellos, el de Linus Pauling y sus colegas, formuló un modelo equivocado, en el cual la molécula de ADN debía estar formada por una triple hélice.
En el segundo equipo, liderado por Maurice Wilkins, trabajaba Rosalind Franklin. Ella fue la primera en obtener una excelente fotografía del ADN por difracción de rayos X, a partir de la cual podía deducirse la distribución y la distancia entre los átomos que formaban parte del ADN. Cuentan que mientras Wilkins y Franklin intentaban traducir sus datos en una estructura probable, la fotografía fue vista por James Watson y Francis Crick, el tercer equipo que estaba investigando la estructura del ADN.

Watson y Crick tenían en mente una serie de posibles estructuras, pero al carecer de buenas fotografías no podían concluir sobre cuál era la correcta. La fotografía de Franklin fue clave en este sentido, y así Watson y Crick pudieron publicar en 1953, en el mismo número de la revista Nature en el que publicaron sus fotografías Wilkins y Franklin, la estructura de doble hélice del ADN. Watson y Crick inician su artículo original de esta manera:
“Deseamos sugerir una estructura para el ácido desoxirribonucleico (ADN). Esta estructura tiene características novedosas que son de considerable interés desde el punto de vista biológico”.
Según el modelo de Watson y Crick, el ADN debía ser una doble hélice y calcularon las distancias exactas que debía haber entre las cadenas y entre los átomos que las componen. Dedujeron que una pirimidina siempre se enfrentaba a una purina de la otra hebra y que estas bases se unían por puentes de hidrógeno.
La estructura de la doble hélice sin duda revolucionó la biología molecular. Más allá de haber sido validada por una infinidad de experimentos y técnicas, proporcionó respuestas a muchas preguntas que se tenían sobre la herencia. Predijo la autorreplicación del material genético y la idea de que la información genética estaba contenida en la secuencia de las bases.
En 1962 James Watson, Francis Crick y Maurice Wilkins recibieron el premio Nobel en medicina por el descubrimiento de la estructura del ADN. Rosalind Franklin había fallecido en 1958, a los 37 años de edad.
Bibliografía consultada
- Judson, Horace F. The eighth day of creation. Makers of the Revolution in Biology. Cold Spring Harbor Laboratory Press. 1996.
- Stent G & Calender R. Genética Molecular. Ediciones Omega, Barcelona. 1981.
- The double helix - 50 years. Nature. 2003.
2.7. La ingeniería genética
Cuando los científicos comprendieron la estructura de los genes y cómo la información que portaban se traducía en funciones o características, comenzaron a buscar la forma de aislarlos, analizarlos, modificarlos y hasta de transferirlos de un organismo a otro para conferirle una nueva característica. Justamente, de eso se trata la ingeniería genética (también llamada metodología del ADN recombinante), un conjunto de técnicas que permite transferir genes de un organismo a otro. Como consecuencia, la ingeniería genética sirve expresar genes (producir las proteínas para las cuales estos genes codifican) en organismos diferentes al de origen. Así, es posible no sólo obtener las proteínas de interés (“recombinates”), sino también mejorar cultivos y animales. Se ha utilizado la ingeniería genética para producir, por mencionar apenas algunos ejemplos:
- Vacunas, como la de la hepatitis B
- Fármacos, como la insulina y la hormona del crecimiento humano
- Enzimas para disolver manchas, como las que se usan en los detergentes en polvo
- Enzimas para la industria alimenticia, como las empleadas en la elaboración del queso y en la obtención de jugos de fruta.
- Plantas resistentes a enfermedades y herbicidas.
- Peces que crecen más rápido
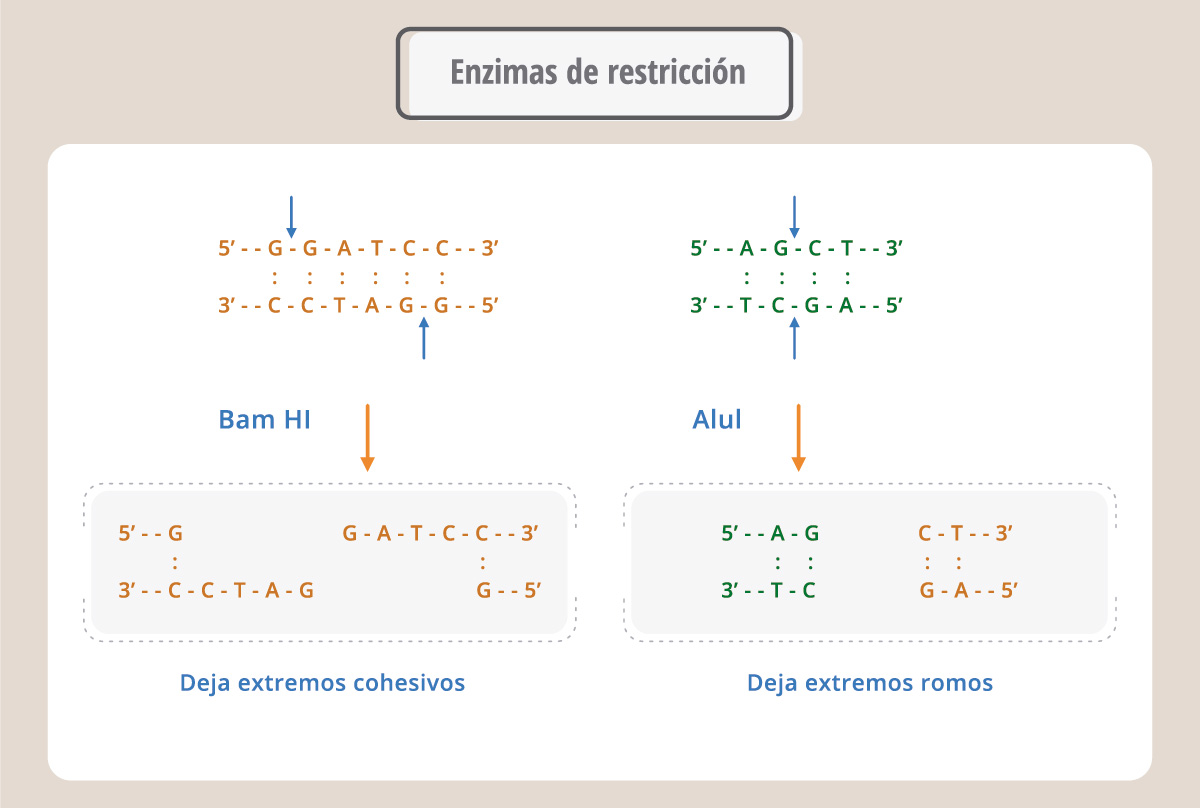
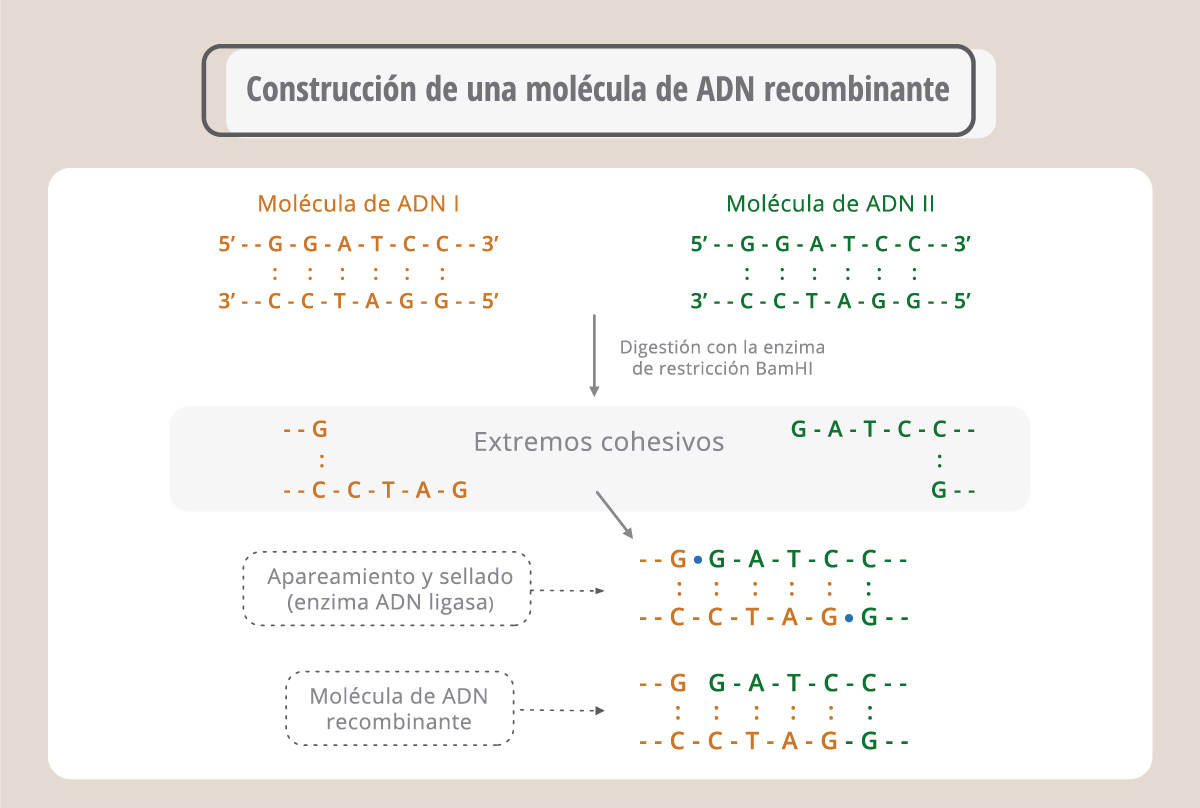
El desarrollo de la ingeniería genética (también llamada metodología del ADN recombinante) fue posible gracias al descubrimiento de las enzimas de restricción y de los plásmidos. Las enzimas de restricción reconocen secuencias determinadas en el ADN. De esta manera, conociendo la secuencia de un fragmento de ADN es posible aislarlo del genoma original para insertarlo en otra molécula de ADN. Hay muchas enzimas de restricción obtenidas a partir de bacterias y que sirven como herramientas para la ingeniería genética. Las enzimas de restricción reconocen secuencias de 4, 6 o más bases y cortan generando extremos romos o extremos cohesivos. Estos extremos, generados en diferentes moléculas de ADN, pueden sellarse con la enzima ADN ligasa y generar así una molécula de ADN nueva, denominada recombinante.
Los plásmidos son moléculas de ADN circulares, originalmente aisladas de bacterias y que pueden extraerse de las mismas e incorporarse a otras, a través del proceso de transformación. Los plásmidos fueron modificados por los investigadores para ser empleados como “vectores”. Así, el gen de interés puede insertarse en el plásmido-vector e incorporarse a una nueva célula. Para seleccionar las células (bacterias o células animales o vegetales) que recibieron el plásmido, éste lleva, además del gen de interés (por ej., el gen de la insulina humana), un gen marcador de selección (por. ej., de resistencia a un antibiótico), que le otorga a la célula que lo lleva la capacidad de sobrevivir en un medio de cultivo selectivo (medio con antibiótico, en este ejemplo). Las células que sobreviven se dividen y generan colonias, formadas por bacterias idénticas. Estas bacterias se denominan recombinantes o genéticamente modificadas. El plásmido recombinante puede aislarse de estas colonias y transferirse a otras células.
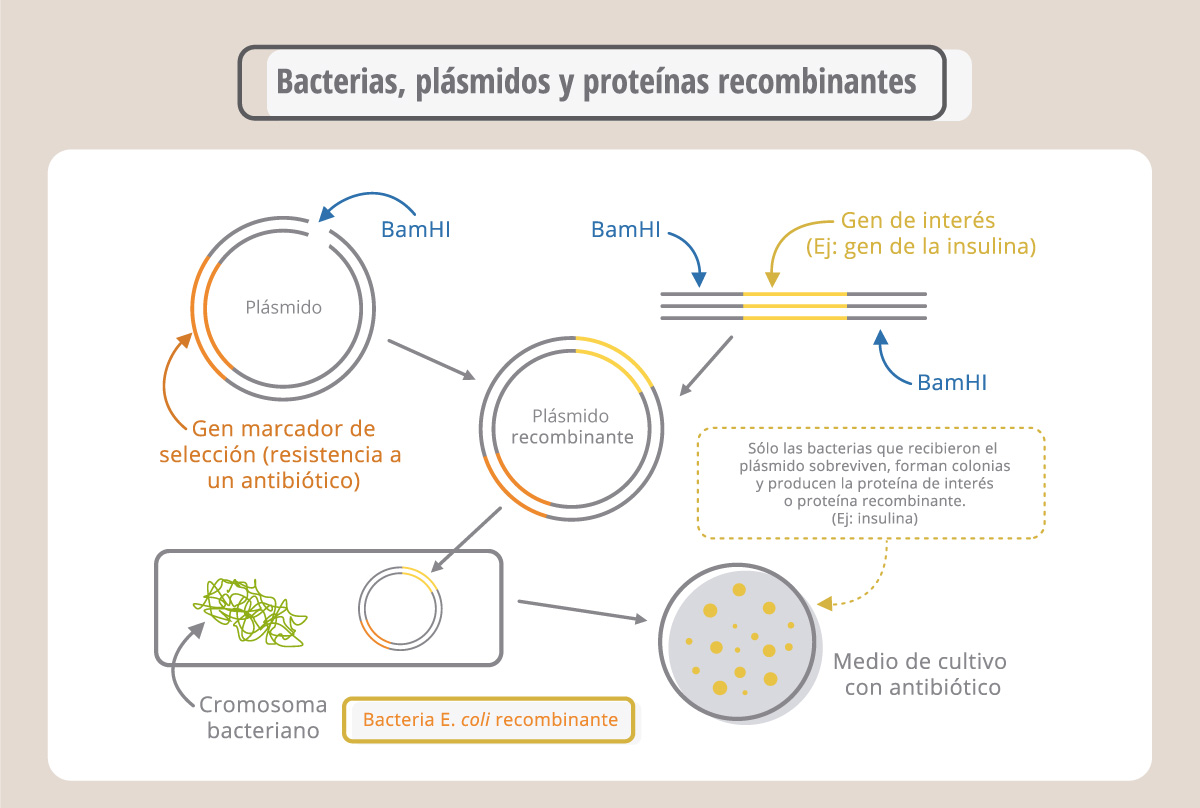
Por esta metodología es posible introducir genes de interés en todo tipo de células, empleando los vectores y las técnicas propias de cada sistema. Podemos entonces generalizar los pasos de la ingeniería genética de la siguiente manera:
- Identificar un carácter o rasgo deseable en el organismo de origen.
- Encontrar el gen responsable del carácter deseado (gen de interés).
- Combinar dicho gen con otros elementos necesarios (vector) para que éste sea funcional en el organismo receptor.
- Transferir el gen de interés, previamente introducido en el vector adecuado, al organismo receptor.
- Crecer y reproducir el organismo receptor, ahora modificado genéticamente.
Por ejemplo, para el caso de la transferencia de un gen insecticida de una bacteria al maíz:
- Identificar la característica “resistencia a insectos” en el organismo de origen, la bacteria del suelo Bacillus thuringiensis.
- Encontrar al gen que lleva las instrucciones para esta característica.
- Combinar este gen con otros elementos genéticos para que sea funcional ahora en una planta (ligarlo a un vector).
- Transferir este gen a células de maíz (organismo receptor).
- Identificar las células de maíz que recibieron el gen (células transformadas) y regenerar, a partir de estas células, una planta adulta.
- Cuaderno para docentes Nº 4: ¿Qué es la ingeniería genética?
2.8. Las "ómicas"
El término “ómicas” hace referencia a las disciplinas como la genómica, la proteómica, la transcriptómica y la metabolómica. A estas tres últimas también se las agrupa bajo la denominación de “genómica funcional”, ya que estudian a los productos de la expresión de los genes. Todas las “ómicas” se basan en el análisis de un gran volumen de datos, y por lo tanto se valen de la bioinformática y de técnicas rápidas y automatizadas de alto rendimiento (high-throughput techniques).
La genómica y la metagenómica
La genómica estudia a los genomas de los organismos. Este estudio incluye la secuenciación del ADN, el análisis de las secuencias para encontrar genes y su comparación con secuencias genómicas de otros organismos. La bioinformática y la tecnología de micromatrices son herramientas fundamentales de la genómica. El primer organismo vivo que tuvo su genoma secuenciado fue la bacteria Haemophilus influenzae, en 1995. A partir de ese momento, y bajo la denominación de “Proyectos Genoma”, se ha completado la secuencia de los genomas de muchas especies más, incluyendo bacterias, hongos, protozoarios, plantas y animales, e incluso del genoma humano.
Algunas de las especies que ya tienen su genoma completamente secuenciado
Especie |
Grupo, denominación común |
Tamaño del genoma (Mpb) |
Finalización del proyecto |
|
Haemophilus influenzae |
Eubacteria (causa la influenza o gripe) |
1,8 |
1995 |
|
Saccharomyces cerevisiae |
Hongo (levadura de cerveza) |
12,07 |
1996 |
|
Bacillus subtilis |
Eubacteria (especie modelo y de uso industrial) |
4,2 |
1997 |
|
Escherichia coli |
Eubacteria (especie modelo y de uso industrial y en ingeniería genética) |
4,6 |
1997 |
|
Caenorhabditis elegans |
Gusano cilíndrico (especie modelo) |
97 |
1998 |
|
Plasmodium falciparum |
Protozoario (causa la malaria) |
23 |
1998 |
|
Helicobacter pylori |
Eubacteria (asociada a úlceras gástricas) |
1,6 |
1999 |
|
Pyrococcus furiosus |
Arquibacteria hipertermófila |
1,9 |
1999 |
|
Homo sapiens |
Hombre |
3.038 |
1999 |
|
Drosophila melanogaster |
Mosca de la fruta (especie modelo) |
180 |
2000 |
|
Vibrio cholerae |
Eubacteria (causa el cólera) |
2,82 |
2000 |
|
Halobacterium sp. |
Arquibacteria (halófila) |
2,59 |
2000 |
|
Arabidopsis thaliana |
Planta (especie modelo) |
119,2 |
2000 |
|
Agrobacterium tumefaciens |
Eubacteria (empleada para transformación genética de plantas) |
5,65 |
2001 |
|
Oryza sativa Japonica |
Planta (arroz) |
389 |
2002 |
|
Candida glabrata |
Hongo (levadura patógena) |
12,28 |
2004 |
|
Entamoeba histolytica |
Protozoario (causa amebiasis intestinal) |
20 |
2004 |
|
Kluyveromyces lactis |
Hongo (levadura de uso industrial) |
10,69 |
2004 |
|
Mus musculus |
Ratón (especie modelo) |
2.500 |
2005 |
|
Trypanosoma cruzi |
Protozoario (causa la Enfermedad de Chagas) |
67 |
2005 |
|
Lactobacillus casei |
Eubacteria (con propiedades probióticas) |
2,93 |
2006 |
|
Pichia stipitis |
Hongo (levadura de uso industrial) |
15,4 |
2007 |
|
Zea mays |
Planta (maíz) |
2.400 |
2008 |
|
Thalassiosira pseudonana |
Diatomea marina (especie modelo) |
34 |
2009 |
- Cuaderno para docentes de PorQué Biotecnología Nº 55: El proyecto Genoma Humano
La metagenómica, también llamada genómica ambiental o genómica de comunidades, es una rama de la genómica en la que se estudian los genomas de comunidades enteras de microbios, sin la necesidad de aislarlos previamente. Esto constituye una gran ventaja, ya que se cree que con los métodos tradicionales, basados en el aislamiento y cultivo previo de los microorganismos, se “pierden” hasta un 99% de los microbios de una muestra. Los proyectos metagenómicos se inician a partir de la toma de una muestra de un ambiente particular (agua, suelo, boca, etc.). Luego se extrae el ADN de la muestra, y se lo secuencia para estudios comparativos o para la búsqueda de genes (funciones) particulares. Hay varios proyectos de este tipo, con objetivos y alcances diferentes. Entre ellos vale la pena destacar el estudio metagenómico de los microorganismos del Mar Sargasso, del drenaje ácido de una mina y otros ambientes extremos, del cuerpo humano y del rumen de bovinos, del suelo, y de los microbios que ayudan al crecimiento de los cultivos o participan en la degradación de la celulosa.
La proteómica
La proteómica estudia y compara cuali- y cuantitativamente el perfil de proteínas (proteoma) presentes en un conjunto de células, tejido u organismo en un momento o condición particular. No sólo se limita a analizar el resultado de la expresión génica, sino que también estudia las modificaciones post-traduccionales que pueden sufrir las proteínas, así como la interacción entre ellas. Las técnicas empleadas son, principalmente, electroforesis en geles bidimensionales, espectrometría de masa y micromatrices o microarreglos de proteínas (animación sobre los métodos empleados)
Se considera a la proteómica como el paso siguiente a la genómica en el estudio de los sistemas biológicos. Mientras el genoma es prácticamente invariable, el proteoma no sólo difiere de célula en célula sino que también cambia según las interacciones bioquímicas con el genoma y el ambiente. Además, las proteínas son más complejas y diversas que los ácidos nucleicos y los genes. Como ejemplo, el genoma humano tiene unos 25.000 genes, y su expresión genera al menos unas 500.000 proteínas diferentes, debido a mecanismos como el splicing alternativo y a modificaciones post-traduccionales.
La transcriptómica
La transcriptómica estudia y compara transcriptomas, es decir, los conjuntos de ARN mensajeros o transcriptos presentes en una célula, tejido u organismo. Como los proteomas, los transcriptomas son muy variables, ya que muestran qué genes se están expresando en un momento dado. Son particularmente interesantes para los científicos los transcriptomas de las células cancerosas y de las células madre, ya que pueden ayudar a entender los complicados procesos de carcinogénesis y de desarrollo y diferenciación celular.
La metabolómica
La metabolómica es el estudio y comparación de los metabolomas, es decir, la colección de todos los metabolitos (moléculas de bajo peso molecular) presentes en una célula, tejido u organismo en un momento dado. Estos metabolitos incluyen a intermediarios del metabolismo, hormonas y otras moléculas de señalización, y a metabolitos secundarios. En 2007, los científicos lograron completar el primer borrador del metaboloma humano. Catalogaron y caracterizaron a unos 2.500 metabolitos, unas 1.200 drogas y unos 3.500 componentes alimenticios que pueden encontrarse en el cuerpo humano. El metaboloma es muy dinámico, cambia ante la menor señal física o química, y debido a que son muchos los tipos de metabolitos que puede haber en una célula, también son varios los métodos que se emplean en el análisis. Para estudiar el metaboloma se necesita primero separar los metabolitos, y luego detectarlos.
Para separarlos se suelen usar las técnicas de cromatografía en fase gaseosa, cromatografía líquida de alto rendimiento (o HPLC), o electroforesis con capilares. Para la detección de los metabolitos, se emplean principalmente la espectrometría de masa y la espectroscopía de resonancia magnética nuclear.
Aunque se usan prácticamente en forma indistinta, para algunos autores los términos metabolómica y metabonómica hacen referencia a objetivos diferentes. Mientras la metabolómica cataloga y cuantifica a las moléculas pequeñas que se encuentran en los sistemas biológicos, la metabonómica estudia cómo cambian los perfiles metabólicos como respuesta a estreses, tales como enfermedades, tóxicos o cambios en la dieta.
Entre las posibles aplicaciones de la metabolómica se encuentran los estudios toxicológicos, ya que se podría estudiar el metaboloma de la orina y otros fluidos corporales para detectar los cambios fisiológicos causados por la exposición a un posible tóxico. Como parte de la genómica funcional, la metabolómica puede ser una herramienta para estudiar la función de los genes, a través de la mutación, deleción o inserción de los mismos. En la nutrigenómica, que relaciona a las “ómicas” con la nutrición humana, la metabolómica podría servir para correlacionar los perfiles de metabolitos de fluidos y órganos con patologías, constitución genética y dietas.


